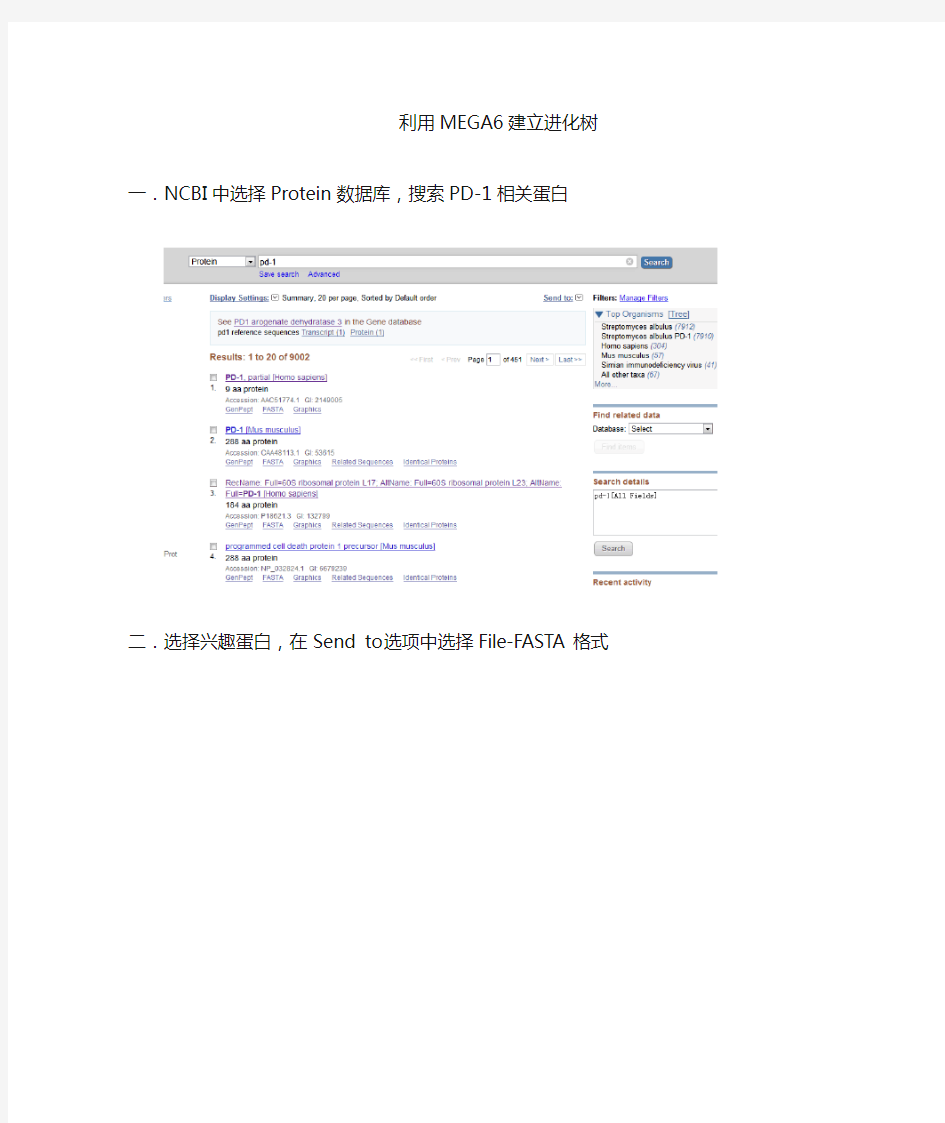

利用MEGA6建立进化树一.NCBI中选择Protein数据库,搜索PD-1相关蛋白
二.选择兴趣蛋白,在Send to选项中选择File-FASTA格式
三.下载安装MEGA6,刚刚下载的FASTA文件变为可读,双击用MEGA6打开
四.展开上图得到下图,至此蛋白序列成功输入MEGA
五.选择菜单栏上Alignment-Align by Clustalw进行蛋白序列比对
六.得到比对后结果,菜单栏-Data-Export Alignment输出比对结果, 得到MEGA6源文件,接下来就可以进行进化树绘制
八.返回主程序界面,菜单栏选择Phylogeny-第二个选项Neighbor,选择刚刚输出的数据,在个性化界面按需要自己的做调整,最后得到所需进化树.
CCS V6.0安装学习流程 ---------write by 聆听忧郁 CCS和IAR相同,都是MSP430的开发工具,不同的是CCS是TI自己研发的。既然如此,CCS对于MSP430的开发一定有一些独到之处吧……最独到的地方是CCS有一个Grace模块,能够进行图形化设置并生成对应代码。单片机内部的模块很多,想快速上手相当困难,Grace模块号称能让新手15分钟就能上手操作,可见其配置简单。 ①下载安装: 这个软件是收费的,但是由于TI大学计划的存在,对于我们来说就相当于免费的……首先用学校邮箱注册TI帐号,已注册的可以更改绑定邮箱,改为学校邮箱,这个验证成功后以后包括申请芯片都会很省事的。因为如果不能认定你是学生,将会收到二次验证,得到的免费服务也会特别少。 注册之后直接搜索CCS,然后点击免费版下载,会弹出一个调查,随便填一下就好,不过一定要合理。
填完之后会收到一封邮件,点一下这个链接就回到下载页面了。 下载很慢……下载的时候要注意的就是要关掉防火墙。 ②安装: 说它是安装,其实是边下载边安装……
然后在安装过程中把和MSP有关的东西全打上钩一路无脑NEXT就好了。 CCS最终程序。 软件和IAR一样,是全英文的。不懂的尽管问谷歌好了…… ③Grace模块安装: 传的神乎其神的Grace竟然是外部模块,这让我无法理解…… 打开CCS。 这个钩最好不要打上,因为路径改起来很困难。 首先找到APP中心。
没下载的这里应该是蓝色的按钮,点选它,然后点上面的INSTALL下载,不过下载速度依然很慢。 ④建立工程: 基本上使用默认参数就好。
大家好: 我在此介绍几个进化树分析及其相关软件的使用和应用范围。这几个软件分别是PHYLIP、PUZZLE、PAUP、TREEVIEW、CLUSTALX和PHYLO-WIN (LINUX)。 在介绍软件之前,我先简要地叙述一下有关进化树分析的一些方法学问题。进化树也称种系树,英文名叫“Phyligenetic tree”。对于一个完整的进化树分析需要以下几个步骤:⑴要对所分析的多序列目标进行排列(To align sequences)。做ALIGNMENT的软件很多,最经常使用的有CLUSTALX和CLUSTALW,前者是在WINDOW下的而后者是在DOS下的。⑵要构建一个进化树(To reconstrut phyligenetic tree)。构建进化树的算法主要分为两类:独立元素法(discrete character methods)和距离依靠法(distance methods)。所谓独立元素法是指进化树的拓扑形状是由序列上的每个碱基/氨基酸的状态决定的(例如:一个序列上可能包含很多的酶切位点,而每个酶切位点的存在与否是由几个碱基的状态决定的,也就是说一个序列碱基的状态决定着它的酶切位点状态,当多个序列进行进化树分析时,进化树的拓扑形状也就由这些碱基的状态决定了)。而距离依靠法是指进化树的拓扑形状由两两序列的进化距离决定的。进化树枝条的长度代表着进化距离。独立元素法包括最大简约性法(Maximum Parsimony methods)和最大可能性法(Maximum Likelihood methods);距离依靠法包括除权配对法(UPGMAM)和邻位相连法(Neighbor-joining)。⑶对进化树进行评估。主要采用Bootstraping法。进化树的构建是一个统计学问题。我们所构建出来的进化树只是对真实的进化关系的评估或者模拟。如果我们采用了一个适当的方法,那么所构建的进化树就会接近真实的“进化树”。模拟的进化树需要一种数学方法来对其进行评估。不同的算法有不同的适用目标。一般来说,最大简约性法适用于符合以下条件的多序列:i 所要比较的序列的碱基差别小,ii 对于序列上的每一个碱基有近似相等的变异率,iii 没有过多的颠换/转换的倾向,iv 所检验的序列的碱基数目较多(大于几千个碱基);用最大可能性法分析序列则不需以上的诸多条件,但是此种方法计算极其耗时。如果分析的序列较多,有可能要花上几天的时间才能计算完毕。UPGMAM(Unweighted pair group method with arithmetic mean)假设在进化过程中所有核苷酸/氨基酸都有相同的变异率,也就
CCSv5 使用教程 1、CCSv5 软件的安装 打开CCSv5的安装包,运行安装包主目录下面的ccs_setup_5.2.1.00018.exe,一路next直到安装完成。(注意:安装包应放在英文目录下) 2、注册破解 首次运行CCSv5需要进行注册,按照软件启动提示或者在help菜单栏下打开注册界面,添加安装包license注册文件,提示注册成功,破解完成。 3、仿真器的安装SEED XDS510PLUS 打开仿真器的驱动安装包,在选择安装目录时选择CCSv5安装目录下的ccs 5.2\ccsv5\ccs_base,将驱动安装在此。 4、项目工程的建立 (1)TI官网下载DSP2833x的标准库文件,安装标准库文件。稍后做库文件的整理。 (2)在你常用的盘符下面建立一个DSP Experiment文件夹,在建立一个名为DSPlib的子文件夹,并在DSPLib文件夹下建立名为Cmd,Lib, Source,Prj 的四个文件夹。 (3)打开标准库的安装目录,在DSP2833x_common和DSP2833x_headers 文件夹下整理出*.cmd存放在Cmd文件夹下,主要有下图的文 件。整理出*.h,*.c,*.asm文件存放在Lib文件夹下。如图。
在Source文件夹下建立main.c的文件。至此建立工程必备的文件都已经准备齐。 (4)(4)在DSP Experiment文件夹下建立example1文件夹,并拷贝Cmd,Lib,Source,Prj 到此文件夹下。然后打开 ccsv5,选择默认的工程项 目文件夹为DSP Experiment,并确定。 (5)Project—>New CCS Project,新建工程,并作如下填写。
Lactobacillus.plantarum 204Lactobacillus.pentosus Lactobacillus.paraplantarum 575Lactobacillus.collinoides Lactobacillus.brevis Lactobacillus.farciminis Lactobacillus.alimentarius Lactobacillus.paralimentarius Lactobacillus.kimchii Lactobacillus.sanfranciscensis Lactobacillus.lindneri Lactobacillus.fructivorans Lactobacillus.hilgardii Lactobacillus.parakefiri Lactobacillus.buchneri Lactobacillus.parabuchneri Lactobacillus.kefiri Lactobacillus.kunkeei P.selangorensis Lactobacillus.perolens Lactobacillus.algidus Lactobacillus.mali Lactobacillus.nagelii Lactobacillus.murinus Lactobacillus.animalis Lactobacillus.ruminus Lactobacillus.equi Lactobacillus.agilis Lactobacillus.cypricasei Lactobacillus.acidipiscis Lactobacillus.salivarius Lactobacillus.salicinius Lactobacillus.aviarius Lactobacillus.araffinosus Lactobacillus.coryniformis Lactobacillus.bifermentans Lactobacillus.sakei Lactobacillus.curvatus Lactobacillus.sharpeae Lactobacillus.manihotivorans Lactobacillus.rhamnosus Lactobacillus.zeae Lactobacillus.casei Lactobacillus.panis Lactobacillus.frumenti Lactobacillus.oris Lactobacillus.vaginalis Lactobacillus.pontis Lactobacillus.reuteri Lactobacillus.colehominis Lactobacillus.mucosae Lactobacillus.fermentum Lactobacillus.amylophilus Lactobacillus.johnsonii Lactobacillus.gasseri Lactobacillus.iners Lactobacillus.jensenii Lactobacillus.fornicalis Lactobacillus.psittaci https://www.doczj.com/doc/556016580.html,ctis Lactobacillus.delbrueckii Lactobacillus.bulgaricus Lactobacillus.acetotolerans Lactobacillus.hamsteri Lactobacillus.amylolyticus Lactobacillus.intestinalis Lactobacillus.gallinarum Lactobacillus.helveticus Lactobacillus.acidophilus Lactobacillus.crispatus Lactobacillus.amylovorus Lactobacillus.fructosus B.subtilis 99579999 99 704924 98 90 79 999999859996949999 9955 99 85746473999985 999445 404332 67 89 7599 998475999972 6599 5799 52 4798 92 97 91853836481621 59 49 3943 358829 37 12 16 0.01
1. 下载 方式多种多样,本机的软件来源于CCS巴的吧友精华帖,一般非特别官方版权的软件都可以在其软件贴吧论坛能找到资源。 本机下载的是CCS勺最新版本32位软件。 2. 安装步骤 a)下载完成得到2个解压包以及一个lie文件,此处我们安装的是 b)双击进入安装界面,首先会弹出两个窗口,分别提示我们:是否同意检测杀毒软件的存在,和杀毒软件正在运行、是否继续安装。 需要注意的是电脑里面的防病毒软件,如果没有关的话会有如下对话窗,这里建议在安装过程中暂时关闭,如果没有关闭的话,引起安装 失败,请看最后的附录 e)同意安装协议 d)选择安装目录,为了规避风险,请在选择安装目录时尽量避免有中文的目录,安装在C盘底下无疑可以运行的更方便,但此处选用其他 功能盘。 e)选择型号 勾选不同的类别就对应着此次安装的内容,安装的过多的内核类别,会导致运算量增加、软件运行变慢,因此需要对照右侧的 Descriptio n,选择我们需要的类别。 本学期的课程围绕C2000的应用,勾选如图选项足以。就算以后需要拓宽我们的业务范围,也可后续在设置中加入,无须担心。 f)这里选择仿真器,一般可以直接安装ccS隹荐的安装就行,如果是有需要,则根据实际情况进行选择安装,单击Next g)这里是CCSV开始拥有的App Center,根据实际需要进行选择,最后单击Finishing。 h)进度条安装等待完成。 3. 启动及配置 a)找到你的安装目录下,双击ccstudio.exe进入初始化界面。 b)选择的工作区间的放置地址,此处我选择新建根目录下的空白文件。 c)正式进入软件界面,弹出一个对话框,询问是否联网下载刚才我们选中的APP STOR内插件,选择NO。 d)安装我们的license注册列表。 选择Upgrand 的Launch license Setup e)找寻我们最初解压出来的那个license文件,最好把他拖到当前安装目录的文件夹下。 点击Browse选择CCS6_License.Li文件。 安装注册表成功的标志是,原本出现在左下角的Free Lice nse变成在 右下角的Full Licensa f)至此,所有安装完成,可以享用美味大餐啦! 对了,不要忘记创建快捷方式,再拖到到桌面啊A _八,此版本不能在桌面出现快捷方式,也无法在开始菜单中自动添加启动方式,需 要手动。 4. 附录
1.MEGA构建系统进化树的步骤 2.CLUSTALX进行序列比对 1.MEGA构建系统进化树的步骤 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。如图: 2. 打开MEGA软件,选择"Alignment" - "Alignment Explorer/CLUSTAL",在对话框中选择Retrieve sequences from a file, 然后点OK,找到准备好的序列文件并打开,如图: 。 3. 在打开的窗口中选择”Alignment”-“Align by ClustalX” 进行对齐,对齐过程需要一段时间,对齐完成后,最好将序列两端切齐,选择两端不齐的部分,
单击右键,选择delete即可,如图: 。 4. 关闭当前窗口,关闭的时候会提示两次否保存,第一次无所谓,保存不保存都可以,第二次一定要保存,保存的文件格式是.meg。根据提示输入Title,然后会出现一个对话框询问是否是Protein-coding nucleotide sequence data, 根据情况选择Yes或No。最后出现一个对话框询问是否打开,选择Yes,如图: 。 5. 回到MEGA主窗口,在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” -“Neighbor-joining”,打开一个窗口,里面有很多参数可以设
置,如何设置这些参数请参考详细的MEGA说明书,不会设置就暂且使用默认值,不要修改,点击下面的Compute按钮,系统进化树就画出来了,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Minimun-evolution”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“Maximun-parsimony”,如图: 在菜单栏中选择”Phylogeny”-“Bootstrap Test of Phylogeny” –“UPGMA”,
目录 第二章软件的安装与应用 (1) 2.1 CCSv5.1的安装 (1) 2.2 利用CCSv5.1导入已有工程 (4) 2.3 利用CCSv5.1新建工程 (6) 2.4 利用CCSv5.1调试工程 (9) 2.5 CCSv5.1资源管理器介绍及应用 (16) 第二章软件的安装与应用 CCS(Code Composer Studio)是TI公司研发的一款具有环境配置、源文件编辑、程序调试、跟踪和分析等功能的集成开发环境,能够帮助用户在一个软件环境下完成编辑、编译、链接、调试和数据分析等工作。CCSv5.1为CCS软件的最新版本,功能更强大、性能更稳定、可用性更高,是MSP430软件开发的理想工具。 2.1 CCSv5.1的安装 (1)运行下载的安装程序ccs_setup_5.1.1.00031.exe,当运行到如图2.1处时,选择Custom 选项,进入手动选择安装通道。 图2.1 安装过程1 (2)单击Next得到如图2.2所示的窗口,为了安装快捷,在此只选择支持MSP430 Low Power MCUs的选项。单击Next,保持默认配置,继续安装。
图2.2 安装过程2 图2.3 软件安装中
图2.4 软件安装完成 (3)单击Finish,将运行CCS,弹出如图2.5所示窗口,打开“我的电脑”,在某一磁盘下,创建以下文件夹路径:-\MSP-EXP430F5529\Workspace,单击Browse,将工作区间链接到所建文件夹,不勾选"Use this as the default and do not ask again"。 图2.5 Workspace选择窗口 (4)单击OK,第一次运行CCS需进行软件许可的选择,如图2.6所示。 在此,选择CODE SIZE LIMITED(MSP430)选项,在该选项下,对于MSP430,CCS免费开放16KB的程序空间;若您有软件许可,可以参考以下链接进行软件许可的认证:https://www.doczj.com/doc/556016580.html,/index.php/GSG:CCSv5_Running_for_the_first_time,单击Finish即可进入CCSv5.1 软件开发集成环境,如图2.7所示。
1.准备序列文件 准备fasta格式序列文件(fasta格式:大于号>后紧跟序列名,换行后是序列。举例如下)。每条序列可以单独为一个文件,也可以把所有序列放在同一文件内。 核酸序列: >sequence1_name CCTGGCTCAGGATGAACGCT 氨基酸序列: >sequence2_name MQSPINSFKKALAEGRTQIGF 2.多序列比对 打开MEGA 5,点击Align,选择Edit/Build Alignment,选择Create a new alignment,点击OK。
这时需要选择序列类型,核酸(DNA)或氨基酸(Protein)。 选择之后,在弹出的窗口中直接Ctrl + V粘贴序列(如果所有序列在同一个文件中,即可全选序列,复制)。也可以:点击Edit,选择Insert Sequence From File,选择序列文件(可多选)。
序列文件加载之后,呈蓝色背景(为选中状态)。点击按钮,选择Align DNA (如果是氨基酸序列,则会出现Align Protein)。弹出的窗口中设置比对参数,一般都是采用默认参数即可。点击OK,开始多序列比对。
比对完成后,呈现以下状态。 这时需要截齐两端含有---的序列:选中含有---的序列,按键Delete删除(注意:两端都需要截齐)。截齐之后,保存文件为:filename.mas
3.构建系统进化树 多序列比对窗口,点击Data,选择Phylogenetic Analysis,弹出窗口询问:所用序列是否编码蛋白质,根据实际情况选择Yes或No。此时,多序列比对文件就激活了,可以返回MEGA 5主界面建树了。
使用CCS进行DSP编程(一) ——CCS编程入门 TI公司提供了高效的C编译器和集成开发环境Code Composer Studio,学习 (6) 的编程应该从学习CCS的使用开始。 首先安装CCS,CCS的安装有详细的说明,并配有简短的Quick Time的多媒体介绍,对于没有购买CCS的用户,可以从TI处得到30天的试用版(没有硬件仿真功能)。 使用CCS前需要对CCS进行设置,以Simulator为例,运行Setup CCS C6000 1.20,安装Device Driver,对于有硬件支持的仿真器,可以选择配套的CCS驱动,设置完成的画面如下图所示:用户的界面大致相同。 接下来就可以运行CCS了,CCS提供了比较好的例子,对于初学者,仔细学习这些例子,会起到事半功倍的效果。在CCS的Help菜单的Tutorial子菜单下,给出了四个教程,分别是:Code Composer Studio Tutorial、Advanced DSP/BIOS Tutorial、Compiler Tutorial和RTDX Tutorial,用户可以从简单的CCS功能 开始,如创建一个工程文件Project,到创建一个完善的用户程序一步一步的进行。
下面是Code Composer Studio Tutorial的例子: 分别从生成一个简单的“Hello World”程序,到使用DSP/BIOS功能,到程序的调试,实时分析,I/O操作等分6课来讲解,可以领略TI的CCS的强大功能。 下面以“Hello World”程序为例讲一下CCS的使用。 首先打开一个Project文件
使用CCS 6新建TMS320F28335工程 本例程以CCS 6.0下新建TMS320F28335工程为例。用CCS 5,或者新建TI的其它处理器工程的区别也不大,参考本例即可。 所建工程能够实现完全的可移植,即任意拷贝到其他位置或其他电脑都能够直接编译运行,非常方便。 一、CCS 6.0安装
一路Next即可,在处理器支持页面,选择C2000系列即可。根据需要选择。
仿真器也是根据实际选择,XDS100是TI官方的,便宜又实在。
跳出是否安装的页面,均选择安装。 安装完成后,CCS5的大学版的license能够继续用。将其复制到C:\ti\ccsv6\ccs_base\DebugServer\license 即可。 打开软件后,会跳出页面要求设置Workspace路径,注意不能有中文。 Workspace路径也可以更改,如下即可:
二、新建TMS320F28335工程 1)在Workspace文件下,建立Project format for CCS6文件夹,这个文件夹就是我们接下来要建立的工程模板文件夹,需要移植或者拷贝程序,拷贝该文件夹即可。 在Project format for CCS6文件夹下,新建Project文件夹,该文件夹用于存放我们的工程文件。 拷贝以下四个文件夹到Project format for CCS6文件夹下。 DSP2833x_common DSP2833x_headers FPUfastRTS IQmath 注:这四个文件夹可以从TI官方文件得到,需要安装 controlSUITE,TI官网可免费下载。 安装好controlSUITE之后,可以在 C:\ti\controlSUITE\libs\math\ C:\ti\controlSUITE\device_support\f2833x\v141\ 中找到上面的四个文件夹。 (v141为版本号,实际不一定是这个版本,可能有更新)
MEGA构建系统进化树的步骤(以MEGA7为例) 本文是看中国慕课山东大学生物信息学课程总结出来的 分子进化的研究对象是核酸和蛋白质序列。研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA 序列。因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。所以这种情况下应该选用DNA序列,而不选蛋白质序列。2)如果DNA序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。 想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。所以我们以后者为例。 2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。如果是比对好的多序列比对可以直接选择“Analyze”。 3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这里选择熟悉的ClustalW),弹出窗口询问“Nothing selected for alignment,Select all?”选择“OK”。 4. 之后,弹出多序列比对参数设置窗口。这个窗口和EMBL在线多序列比对一样,可以设置替换记分矩阵、不同的空位罚分(罚分填写的是正数,计算时按负数计算)等参数。MEGA的所有默认参数都是经过反复考量设置的,这保证了MEGA傻瓜机全自动档的品质,所以当你无从下手,或者没有什么特别要求的时候,直接点击“OK”,接受这些默认参数,开始多序列比对。
第一步:打开软件 下面介绍菜单的使用: Data菜单: Creat a new :创建一个新的数据比对文件,也就是说当我们比对完一组后,想接着比对另一组,那么使用它就可以不用退出直接把数据文件导入; Open :打开先前已经比对并保存好的文件,它包含两个子菜单:retive sequence from file 和saved aligment session ; Close: 关闭当前的比对数据文件;
Save session :保存当前比对结果,可以给比对的结果一个文件名; Export alignment :将当前的序列比对结果输出到指定文件,有两种输入格式可供选 择:MGTA 和FASTA. DNA sequence :使用它来选择输入的数据DNA 序列,这里需要说明的是如果你输入的数据是氨基酸序列的话,比对窗口只显示一个标签,若是DNA 序列的话则显示两个标签,一个是DNA 序列的,另一个是氨基酸序列的。 Protein sequences :选择输入的氨基酸序列,选择后,所以的位点就被当作氨基酸残 基位点来对待。 Translate/untranslate :只有比对的序列是编码蛋白的DNA序列的时候才可用。它可以根据指定的遗传密码表将DNA 序列翻译成特定的氨基酸序列。 Select genetic code table :使用它将编码蛋白的DNA 翻译成特定的蛋白序列。 R everse complement :将选择的一整行的DNA 序列变为与之互补配对碱基序列。Exit alignment explorer :退出序列比对的资源管理窗口 Edit 菜单: 使用这个菜单可以对我们的比对序列进行想要的一些编辑工作具体为 Undo:撤销上一步操作; Copy:复制;Cut:剪切;Paste:粘贴;这三个操作都可以只针对一个碱基或 氨基酸残基也可以是一段甚至是整个序列; Delete:从比对表格中删除一段序列; Delete gaps:去掉序列中的空缺; Insert blank sequence:重新插入一空行;标签和序列都是空的; Insert sequence from file :从已保存的文件中插入新的序列;
构建系统进化树的方法步骤 1. 建树前的准备工作 1.1 相似序列的获得——BLAST BLAST是目前常用的数据库搜索程序,它是Basic Local Alignment Search Tool的缩写,意为“基本局部相似性比对搜索工具”(Altschul et al.,1990[62];1997[63])。国际著名生物信息中心都提供基于Web的BLAST服务器。BLAST算法的基本思路是首先找出检测序列和目标序列之间相似性程度最高的片段,并作为内核向两端延伸,以找出尽可能长的相似序列片段。 首先登录到提供BLAST服务的常用网站,比如国内的CBI、美国的NCBI、欧洲的EBI和日本的DDBJ。这些网站提供的BLAST服务在界面上差不多,但所用的程序有所差异。它们都有一个大的文本框,用于粘贴需要搜索的序列。把序列以FASTA格式(即第一行为说明行,以“>”符号开始,后面是序列的名称、说明等,其中“>”是必需的,名称及说明等可以是任意形式,换行之后是序列)粘贴到那个大的文本框,选择合适的BLAST程序和数据库,就可以开始搜索了。如果是DNA序列,一般选择BLASTN搜索DNA数据库。 这里以NCBI为例。登录NCBI主页-点击BLAST-点击Nucleotide-nucleotide BLAST (blastn)-在Search文本框中粘贴检测序列-点击BLAST!-点击Format-得到result of BLAST。 BLASTN结果如何分析(参数意义): >gi|28171832|gb|AY155203.1| Nocardia sp. ATCC 49872 16S ribosomal RNA gene, complete sequence Score = 2020 bits (1019), Expect = 0.0 Identities = 1382/1497 (92%), Gaps = 8/1497 (0%) Strand = Plus / Plus Query: 1 gacgaacgctggcggcgtgcttaacacatgcaagtcgagcggaaaggccctttcgggggt 60 |||||||||||||||||||||||||||||||||||||||||| ||||||||| ||||| Sbjct: 1 gacgaacgctggcggcgtgcttaacacatgcaagtcgagcggtaaggcccttc--ggggt 58 Query: 61 actcgagcggcgaacgggtgagtaacacgtgggtaacctgccttcagctctgggataagc 120 || ||||||||||||||||||||||||||||||| | |||||| ||||||||||||| Sbjct: 59 acacgagcggcgaacgggtgagtaacacgtgggtgatctgcctcgtactctgggataagc 118 Score :指的是提交的序列和搜索出的序列之间的分值,越高说明越相似;
如何用MEGA构建进化树 MEGA3、1就是一个关于序列分析以及比较统计得工具包,其中包括有距离建树法与MP建树法;可自动或手动进行序列比对,推断进化树,估算分子进化率,进行进化假设测验,还能联机得Web数据库检索。下载后可直接使用,主要包括几个方面得功能软件:i)DNA与蛋白质序列数据得分析软件。ii)序列数据转变成距离数据后,对距离数据分析得软件。iii)对基因频率与连续得元素分析得软件。iv)把序列得每个碱基/氨基酸独立瞧待(碱基/氨基酸只有0与1得状态)时,对序列进行分析得软件。v)绘制与修改进化树得软件,进行网上blast搜索。 用MEGA构建进化树有以下步骤: 1、16S rDNA测序与参考序列选取 从环境中分离到单克隆,去重复后扩增16S rDNA序列并测序,然后与数据库比对,找到相似度最高得几个序列,确定一下您分离得细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定您分离得到得就就是Blast到得那个,然后找一到两个同科得,再找一到两个同目得,再找一到两个同纲得细菌,把序列全部下下来,以FSATA形式整合在TXT文档中,如 >TS1 GCAGTCGAACGATGAAGCCCAGCTTGCTGGGTGGA TTAGTGGCGAACGGGTGAGTAACACGTGGGTGATCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAATACCGGATAGGACCTCGGGA TGCATGTTCCGGGGTGGAAAGGTTTTCCGGTGCAGGATGGGCC >gi|117572706|gb|EF028124、1| Rhodococcus sp、Atl25 16S ribosomal RNAgene,partial sequence CGATTAGAGTTTGATCCTGGCTCAGGACGAACGCTGGCGGCGTGCTTAACACATGCAAGTCGAACGATGAAGCCCAGCTTGCTGGGTGGATTAGTGGCGAACGGGTGAGTAACACGTGGGTGA TCTGCCCTGCACTTCGGGATAAGCCTGGGAAACTGGGTCTAATACCGGAT >TS2 TGCAAGTCGAGCGAATGGA TTAAGAGCTTGCTCTTATGAAGTTAGCGGCGGA CGGGTGAGTAACACGTGGGTAACCTGCCCATAAGACTGGGATAACTCCGG GAAACCGGGGCTAATACCGGATAACATTTTGAACTGCATGGTTCGAAATTGAAAGGCGGCTTCGGCTGTCACT >gi|56383044|emb|AJ809498、1|Bacillus cereus partial16S rRNA gene, strainTMW 2、383 GA TGAACGCTGGCGGCGTGCCTAATACATGCAAGTCGAGCGAATGGATTAAGAGCTTGCTCTTATGAAGTTAGCGGCGGACGGGTGAGTAACACGTGGGTAACCTGCCCATAAGACTGGGA TAACTCCGGGAAACCGGGGCTAA TACCGGATAACA TTTTGAACYGCA TGGTTC…………………………、 …………………………、 参考序列选择有几个原则:a,不选非培养(unclutured)微生物为参比;b,所选参考序列要正确,里面无错误碱基;c,在保证同属得前提下,优先选择16S rDNA全长测序或全基因组测序得种;d,每个种属选择一个参考序列,如果自己得序列中同一属得较多,可适当选择两个参考序列。 2、序列比对
M E G A构建系统进化树的步骤(以M E G A7为 例)
MEGA构建系统进化树的步骤(以MEGA7为例) 本文是看中国慕课山东大学生物信息学课程总结出来的 分子进化的研究对象是核酸和蛋白质序列。研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA序列。因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。所以这种情况下应该选用DNA序列,而不选蛋白质序列。2)如果DNA 序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。 1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致 ( 5’-3’)。 想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。所以我们以后者为例。 2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。如果是比对好的多序列比对可以直接选择“Analyze”。 3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这
一步一步教你如何做系统进化树 在此介绍几个进化树分析及其相关软件的使用和应用范围。这几个软件分别是PHYLIP 、PUZZLE 、PAUP 、TREEVIEW 、CLUSTALX 和PHYLO-WIN (LINUX )。 在介绍软件之前,我先简要地叙述一下有关进化树分析的一些方法学问题。 进化树也称种系树,英文名叫“Phyligenetic tree ”。对于一个完整的进化树分析需要以下几个步骤:⑴ 要对所分析的多序列目标进行排列(To align sequences )。做ALIGNMENT 的软件很多,最经常使用的有CLUSTALX 和CLUSTALW ,前者是在WINDOW 下的而后者是在DOS 下的。⑵ 要构建一个进化树(To reconstrut phyligenetic tree )。构建进化树的算法主要分为两类:独立元素法(discrete character methods )和距离依靠法(distance methods )。所谓独立元素法是指进化树的拓扑形状是由序列上的每个碱基/氨基酸的状态决定的(例如:一个序列上可能包含很多的酶切位点,而每个酶切位点的存在与否是由几个碱基的状态决定的,也就是说一个序列碱基的状态决定着它的酶切位点状态,当多个序列进行进化树分析时,进化树的拓扑形状也就由这些碱基的状态决定了)。而距离依靠法是指进化树的拓扑形状由两两序列的进化距离决定的。进化树枝条的长度代表着进化距离。独立元素法包括最大简约性法(Maximum Parsimony methods )和最大可能性法(Maximum Likelihood methods );距离依靠法包括除权配对法(UPGMAM )和邻位相连法(Neighbor-joining )。⑶ 对进化树进行评估。主要采用Bootstraping 法。进化树的构建是一个统计学问题。我们所构建出来的进化树只是对真实的进化关系的评估或者模拟。如果我们采用了一个适当的方法,那么所构建的进化树就会接近真实的“进化树”。模拟的进化树需要一种数学方法来对其进行评估。不同的算法有不同的适用目标。一般来说,最大简约性法适用于符合以下条件的多序列:i 所要比较的序列的碱基差别小,ii 对于序列上的每一个碱基有近似相等的变异率,iii 没有过多的颠换/转换的倾向,iv 所检验的序列的碱基数目较多(大于几千个碱基);用最大可能性法分析序列则不需以上的诸多条件,但是此种方法计算极其耗时。如果分析的序列较多,有可能要花上几天的时间才能计算完毕。UPGMAM (Unweighted pair group method with arithmetic mean )假设在进化过程中所有核苷酸/氨基酸都有相同的变异率,也就是存在着一个分子钟。这种算法得到的进化树相对来说不是很准确,现在已经很少使用。邻位相连法是一个经常被使用的算法,它构建的进化树相对准确,而且计算快捷。其缺点是序列上的所有位点都被同等对待,而且,所分析的序列的进化距离不能太大。另外,需要特别指出的是对于一些特定多序列对象来说可能没有任何一个现存算法非常适合它。最好是我们来发展一个更好的算法来解决它。但无疑这是非常难的。我想如果有人能建立这样一个算法的话,那他(她)完全可以在 生 物秀-专心做生物 w w w .b b i o o .c o m
CCS6.0显示AD波形 利用CCS6.0 实时显示两路AD转换结果,用波形的显示的设置步骤如下:1、打开CCS6.0, 打开AD工程,如图:
程序如下: #include "myapp.h" #include "csedu.h" #include "scancode.h" void InitADC(); void wait( unsigned int cycles ); void EnableAPLL( ); unsigned int nADC0[256],nADC1[256]; main() { int i; unsigned int uWork; EnableAPLL(); SDRAM_init(); InitADC(); PLL_Init(132); while ( 1 ) { for ( i=0;i<256;i++ ) { ADCCTL=0x8000; // 启动AD转换,通道0 do { uWork=ADCDATA; } while ( uWork&0x8000 ); nADC0=uWork&0x0fff; }
for ( i=0;i<256;i++ ) { ADCCTL=0x9000; // 启动AD转换,通道1 do { uWork=ADCDATA; } while ( uWork&0x8000 ); nADC1=uWork&0x0fff; } asm( " nop"); // break point } } void InitADC() { ADCCLKCTL=0x23; // 4MHz ADCLK ADCCLKDIV=0x4f00; } void wait( unsigned int cycles ) { int i; for ( i = 0 ; i < cycles ; i++ ){ } } void EnableAPLL( ) { *( ioport volatile unsigned short* )0x1f00 = 4; wait( 25 );
1.下载 方式多种多样,本机的软件来源于CCS吧的吧友精华帖,一般非特别官方版权的软件都可以在其软件贴吧论坛能找到资源。 本机下载的是CCS的最新版本32位软件。 2.安装步骤 a)下载完成得到2个解压包以及一个lic文件,此处我们安装的是 b)双击进入安装界面,首先会弹出两个窗口,分别提示我们:是否同 意检测杀毒软件的存在,和杀毒软件正在运行、是否继续安装。 需要注意的是电脑里面的防病毒软件,如果没有关的话会有如下对话窗,这里建议在安装过程中暂时关闭,如果没有关闭的话,引 起安装失败,请看最后的附录 c)同意安装协议 d)选择安装目录,为了规避风险,请在选择安装目录时尽量避免有中 文的目录,安装在C盘底下无疑可以运行的更方便,但此处选用其 他功能盘。 e)选择型号 勾选不同的类别就对应着此次安装的内容,安装的过多的内核类别,会导致运算量增加、软件运行变慢,因此需要对照右侧的 Description,选择我们需要的类别。 本学期的课程围绕C2000的应用,勾选如图选项足以。就算以后需要拓宽我们的业务范围,也可后续在设置中加入,无须担心。 f)这里选择仿真器,一般可以直接安装CCS推荐的安装就行,如果是 有需要,则根据实际情况进行选择安装,单击Next g)这里是CCSV开始拥有的App Center,根据实际需要进行选择,最后 单击Finishing。 h)进度条安装 等待完成。 3.启动及配置 a)找到你的安装目录下F:\,双击ccstudio.exe进入初始化界面。 b)选择的工作区间的放置地址,此处我选择新建根目录下的空白文件。 c)正式进入软件界面,弹出一个对话框,询问是否联网下载刚才我们选 中的APP STORE内插件,选择NO。 d)安装我们的license注册列表。 选择Upgrand的Launch license Setup。 e)找寻我们最初解压出来的那个license文件,最好把他拖到当前安装 目录的文件夹下。 点击Browse,选择CCS6_License.Lic文件。 安装注册表成功的标志是,原本出现在左下角的Free License变成在 右下角的Full License。 f)至此,所有安装完成,可以享用美味大餐啦! 对了,不要忘记创建快捷方式,再拖到到桌面啊^ _ ^,此版本不能在桌面出现快捷方式,也无法在开始菜单中自动添加启动方式,需 要手动。 4.附录