
.. 一、常规基因敲除鼠(Conventional Knockout) 常规基因敲除是通过基因打靶,把需要敲除的基因的几个重要的外显子或者功能区域用Neo Cassette 替换掉。这样的小鼠其全身所有的组织和细胞中都不表达该基因产物。此类基因敲除鼠一般用于研究某个基因在对小鼠全身生理病理的影响,而且这个基因没有胚胎致死性。 二、条件性基因敲除小鼠(Conditional Knockout) 条件性基因敲除小鼠是通过基因打靶,把两个loxP 位点放到目的基因一个或几个重要的外显子的两边。该小鼠和表达Cre酶小鼠杂交之前,其目的基因表达完全正常。当和组织特异性表达Cre酶的小鼠进行杂交后,可以在特定的组织或细胞中敲除该基因,而该基因在其他组织或细胞表达正常。 条件性基因敲除鼠适用范围为:(1)该基因有胚胎致死性;(2)用于研究该基因在特定的组织或细胞中的生理病理功能。 三、基因敲入小鼠(Knockin) 基因敲入小鼠是通过基因打靶,把目的基因序列敲入到小鼠的相应基因位点,使用小鼠的表达调控元件指导目的基因表达。 此类基因敲入鼠一般用于药物的筛选,信号通路的研究等。 获得嵌合体及之后品系纯化详细流程: 基因敲除其他方法: 一、ZFN技术制作基因敲除鼠 ZFN能够识别并结合指定的基因序列位点,并高效精确地切断。随后细胞利用天然的DNA 修复过程来实现DNA的插入、删除和修改,这样研究人员就能够随心所欲地进行基因组编辑。这在过去是无法想象的,传统的基因敲除技术依赖细胞内自然发生的同源重组,其效率只有百万分之一,而ZFN的基因敲除效率能达到10%。利用这些技术进行小鼠基因的定点敲除和敲入,可以把时间从一年缩短到几个月。 这项技术中设计特异性的ZFN是最关键的环节,目前研究者采用计算生物学方法设计高特异性的ZFN,但ZFN的脱靶(off target),也就是把不该切的地方切了的问题仍是一个挑战。也正因为这个原因,利用ZFN技术进行小鼠的基因修饰还无法完全取代传统技术。 二、TALEN技术制作基因敲除鼠 TALEN 技术是一种崭新的分子生物学工具。科学家发现,来自一种植物细菌的TAL蛋白的核酸结合域的氨基酸序列与其靶位点的核酸序列有恒定的对应关系。利用TAL的序列模块,可组装成特异结合任意DNA序列的模块化蛋白,从而达到靶向操作内源性基因的目的,它克服了ZFN方法不能识别任意目标基因序列,以及识别序列经常受上下游序列影响等问题,而具有ZFN相等或更好的灵活性,使基因操作变得更加简单方便。然而同样因为脱靶的问题,利用TALEN技术进行小鼠的基因修饰仍然无法取代传统技术。 ;.
LentiCRISPRv2 and lentiGuide-Puro: lentiviral CRISPR/Cas9 and single guide RNA CRISPR (C lustered R egularly I nterspaced S hort P alindromic R epeats) is a microbial nuclease system involved in defense against invading phages and plasmids. CRISPR loci in microbial hosts contain a combination of CRISPR-associated (Cas) genes as well as non-coding RNA elements capable of programming the specificity of the CRISPR-mediated nucleic acid cleavage. Lentiviral CRISPR/Cas can infect a broad variety of mammalian cells by co-expressing a mammalian codon-optimized Cas9 nuclease along with a single guide RNA (sgRNA) to facilitate genome editing (Shalem*, Sanjana*, et al., Science 2014). Protocols for cloning into the lentiviral transfer plasmid and general considerations for producing lentivirus are described below. Separate protocols are available for amplifying the genome-scale CRISPR knock-out (GeCKO) libraries. This protocol is for creating individual lentiviral CRISPR plasmids targeting a single genomic locus. lentiCRISPRv2 (one vector system): This plasmid contains two expression cassettes, hSpCas9 and the chimeric guide RNA. The vector can be digested using BsmB I, and a pair of annealed oligos can be cloned into the single guide RNA scaffold. The oligos are designed based on the target site sequence (20bp) and needs to be flanked on the 3' end by a 3bp NGG PAM sequence, as shown on the next page. lentiGuide-Puro (two vector system): This plasmid expressed only the chimeric guide RNA. It does not contain Cas9. Please use lentiCas9-Blast (a separate lentiviral construct that delivers hSpCas9 and blasticidin resistance) to first integrate Cas9 into your cell line. The lentiGuide-Puro vector can be digested using BsmB I, and a pair of annealed oligos can be cloned into the single guide RNA scaffold. The oligos are designed based on the target site sequence (20bp) and needs to be flanked on the 3' end by a 3bp NGG PAM sequence, as shown on the next page. Which vector to use: lentiCRISPRv2 is identical to the original lentiCRISPRv1 but produces nearly 10X higher titer virus. lentiGuide-Puro produces >100X higher titer virus over lentiCRISPRv1 and should be used in cell lines where Cas9 has already been integrated in (e.g. using the separate lentiCas9-Blast lentivirus). For applications where Cas9 cannot first be introduced (e.g. primary cells), lentiCRISPRv2 is recommended. After transduction, use puromycin to select for cells with lentiCRISPRv2 or lentiGuide-Puro. Lentiviral production: Before starting any lentiviral work, please ensure compliance with your Environmental Health and Safety office and government/organization/university. Briefly, to make lentivirus, a transfer plasmid (e.g. lentiCRISPRv2 or lentiGuide-Puro) must be co-transfected into HEK293(F)T cells with the packaging plasmids pVSVg (AddGene 8454) and psPAX2 (AddGene 12260). As a positive control for viral production, we often use a CMV-EGFP lentiviral transfer plasmid (eg. AddGene 19319). Target design notes and online resources: For application of Cas9 for site-specific genome editing in eukaryotic cells and organisms, we have computationally identified suitable target sites for the S. pyogenes Cas9 and calculated most likely off-targets within the genome. Please visit https://www.doczj.com/doc/7f3046973.html, to access these Cas9 target design tools. Complete plasmid sequences, protocols, a discussion forum and additional information can be found at the Zhang Lab GeCKO website: https://www.doczj.com/doc/7f3046973.html,/gecko/ . Citation: Please reference the following publications for the use of this material. Improved lentiviral vectors and genome-wide libraries for CRISPR screening. Sanjana NE*, Shalem O*, Zhang F. Nature Methods (2014). Genome-scale CRISPR-Cas9 knockout screening in human cells. Shalem O*, Sanjana NE*, Hartenian E, Shi X, Scott DA, Mikkelsen T, Heckl D, Ebert BL, Root DE, Doench JG, Zhang F (2014). Science, 343, 83-7. DOI: 10.1126/science.1247005
转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,并逐渐从基础研究实验室转向商业模式,成为一项 高度标准化的新兴产业 一、技术介绍与研究进展 转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,至今已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,在小鼠模型构建方面日趋完善,并且如同剪切酶和抗体等常规分子生物学试剂的制备技术一样,逐渐从基础研究实验室转向商业模式,成为一项高度标准化的新兴产业,催生了数以百计的创新药物和数以千计的优秀文章。尽管如此,传统技术仍然存在一些难以克服的缺陷,如步骤繁琐、周期漫长、成功率低、费用高昂等,而ZFN和TALEN等新技术的出现,或有可能将这一局面彻底改变。
二、同源重组技术原理 基因敲除鼠技术是上世纪80年代中后期基于DNA同源重组的原理发展起来的,Capecchi和Smithies在1987年根据同源重组(homologous recombination)的原理,首次实现了ES的外源基因的定点整合(targeted integration),这一技术称为"基因打靶"(gene targeting)或"基因敲除"(gene knockout),利用这种ES的显微注射就可以制作出基因敲出小鼠(KO Mice: knockout mice);由于这一工作,Capecchi和Smithies 于2007年与Evans分享了诺贝尔医学奖。 同源重组(homologous recombination)定义:是指发生在姐妹染色单体(sister chromatin) 之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。在基因敲除小鼠制作过程中,需要针对目的基因两端特异性片段设计带有相同片段的重组载体,将重组载体导入到胚胎干细胞后外源的重组载体与胚胎干细胞中相同的片段会发生同源重组,如图1所示: 图1.基因敲除鼠制作同源重组原理示意图 三、制作流程 图2.基因敲除鼠制作过程示意图 1. Knockout载体设计与构建
基因敲除技术 点击次数: 2605 发布日期: -5-25 来源: 本站仅供参考, 谢 绝转载, 否则责任自负 1.概述: 基因敲除是自80年代末以来发展起来的一种新型分子生物学技术, 是经过一定的途径使机体特定的基因失活或缺失的技术。一般意义上的基因敲除主要是应用DNA同源重组原理, 用设计的同源片段替代靶基因片段, 从而达到基因敲除的 目的。随着基因敲除技术的发展, 除了同源重组外, 新的原理和技术也逐渐被应用, 比较成功的有基因的插入突变和iRNA, 它们同样能够达到基因敲除的目的。2.实现基因敲除的多种原理和方法: 2.1.利用基因同源重组进行基因敲除 基因敲除是80年代后半期应用DNA同源重组原理发展起来的。80年代初, 胚胎干细胞( ES细胞) 分离和体外培养的成功奠定了基因敲除的技术基础。1985年, 首次证实的哺乳动物细胞中同源重组的存在奠定了基因敲除的理论基础。到 1987年, Thompsson首次建立了完整的ES细胞基因敲除的小鼠模型[1]。直到现在, 运用基因同源重组进行基因敲除依然是构建基因敲除动物模型中最普遍的 使用方法。 2.1.1利用同源重组构建基因敲除动物模型的基本步骤(图1):
图1.基因同源重组法敲除靶基因的基本步骤 a.基因载体的构建: 把目的基因和与细胞内靶基因特异片段同源的DNA 分子 都重组到带有标记基因(如neo 基因, TK 基因等)的载体上, 成为重组载体。基因敲除是为了使某一基因失去其生理功能, 因此一般设计为替换型载体。 b.ES 细胞的获得: 现在基因敲除一般采用是胚胎干细胞, 最常见的是鼠, 而兔, 猪, 鸡等的胚胎干细胞也有使用。常见的鼠的种系是129及其杂合体, 因为这类小鼠具有自发突变形成畸胎瘤和畸胎肉瘤的倾向, 是基因敲除的理想实验动物。而其它遗传背景的胚胎干细胞系也逐渐被发展应用。[2, 3] c.同源重组: 将重组载体经过一定的方式(电穿孔法或显微注射)导入同源的胚胎干细胞(ES cell)中, 使外源DNA与胚胎干细胞基因组中相应部分发生同源重组, 将重组载体中的DNA序列整合到内源基因组中, 从而得以表示。一般地, 显微注射命中率较高, 但技术难度较大, 电穿孔命中率比显微注射低, 但便于使用。[4,5] d.选择筛选已击中的细胞: 由于基因转移的同源重组自然发生率极低, 动物的重组概率为10-2~10-5, 植物的概率为10-4~10-5。因此如何从众多细胞中筛出真正发生了同源重组的胚胎干细胞非常重要。当前常见的方法是正负筛选法( PNS法) , 标记基因的特异位点表示法以及PCR法。其中应用最多的是PNS法。[6]
基因敲除小鼠pc鉴 一、技术介绍与研究进展 敲除动物技术已经/ 基因、基因敲入转该技术从上世成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,原核显史,经典技术如DNA纪七八十年代诞生以来,至今已有近四十年的历 在小鼠模型构建方面日趋微注射、胚胎干细胞显微注射技术一直以来经久不衰,制备技术一样,逐渐从完善,并且如同剪切酶和抗体等常规分子生物学试剂的 催生了数以百基础研究实验室转向商业模式,成为一项高度标准化的新兴产业,然存在一些难以计的创新药物和数以千计的优秀文章。尽管如此,传统技术仍TALEN和费用高昂等,而ZFN克服的缺陷,如步骤繁琐、周期漫长、成功率低、等新技术的出现,或有可能将这一局面彻底改变。二、同源重组技术原理同源重组的原理发展起来的,年代中后期基于DNA基因敲除鼠技术是上世纪80)homologous recombination1987年根据同源重组(在Capecchi和Smithies),这的外源基因的定点整合(EStargeted integration的原理,首次实现了),gene knockout(基因敲除)或gene targeting(基因打靶一技术称为 利用这种ES的显微注射就可以制作出基因敲出小鼠(KO Mice: knockout mice);由于这一工作,Capecchi和Smithies于2007年与Evans分享了诺贝
尔医学奖。 同源重组(homologous recombination)定义:是指发生在姐妹染色单体(sister chromatin) 之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。在基因敲除小鼠制作过程中,需要针对目的基因两端特异性片段设计带有相同片段的重组载体,将重组载体导入到胚胎干细胞后外源的重组载体与胚胎干细胞中相同的片段会发生同源重组,如图1所示: 1.基因敲除鼠制作同源重组原理示意图图制作流程 三、. 基因敲除鼠制作过程示意图图2. 载体设计与构建1. Knockout根据研究项目具体情况和要求把目的基因和与细胞内靶基因特异片段同源的的载体上,成为重
慢病毒载体包装构建过程 原理:慢病毒载体可以将外源基因或外源的shRNA有效地整合到宿主染色体上,从而达到持久性表达目的序列的效果。在感染能力方面可有效地感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞、干细胞等多种类型的细胞,从而达到良好的的基因治疗效果。对于一些较难转染的细胞,如原代细胞、干细胞、不分化的细胞等,使用慢病毒载体,能大大提高目的基因或目的shRNA的转导效率,且目的基因或目的shRNA整合到宿主细胞基因组的几率大大增加,能够比较方便快捷地实现目的基因或目的shRNA的长期、稳定表达。 概念:慢病毒载体是指以人类免疫缺陷病毒-1 (H IV-1) 来源的一种病毒载体,慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息,是慢病毒载体系统的主要组成部分。携带有外源基因的慢病毒载体在慢病毒包装质粒、细胞系的辅助下,经过病毒包装成为有感染力的病毒颗粒,通过感染细胞或活体组织,实现外源基因在细胞或活体组织中表达。 辅助成分:慢病毒载体辅助成分包括:慢病毒包装质粒和可产生病毒颗粒的细胞系。 慢病毒载体包含了包装、转染、稳定整合所需要的遗传信息。慢病毒包装质粒可提供所有的转录并包装RNA 到重组的假病毒载体所需要的所有辅助蛋白。为产生高滴度的病毒颗粒,需要利用表达载体和包装质粒同时共转染细胞,在细胞中进行病毒的包装,包装好的假病毒颗粒分泌到细胞外的培养基中,离心取得上清液后,可以直接用于宿主细胞的感染,目的基因进入到宿主细胞之后,经过反转录,整合到基因组,从而高水平的表达效应分子。 基本原理:慢病毒载体系统由两部分组成,即包装成分和载体成分。
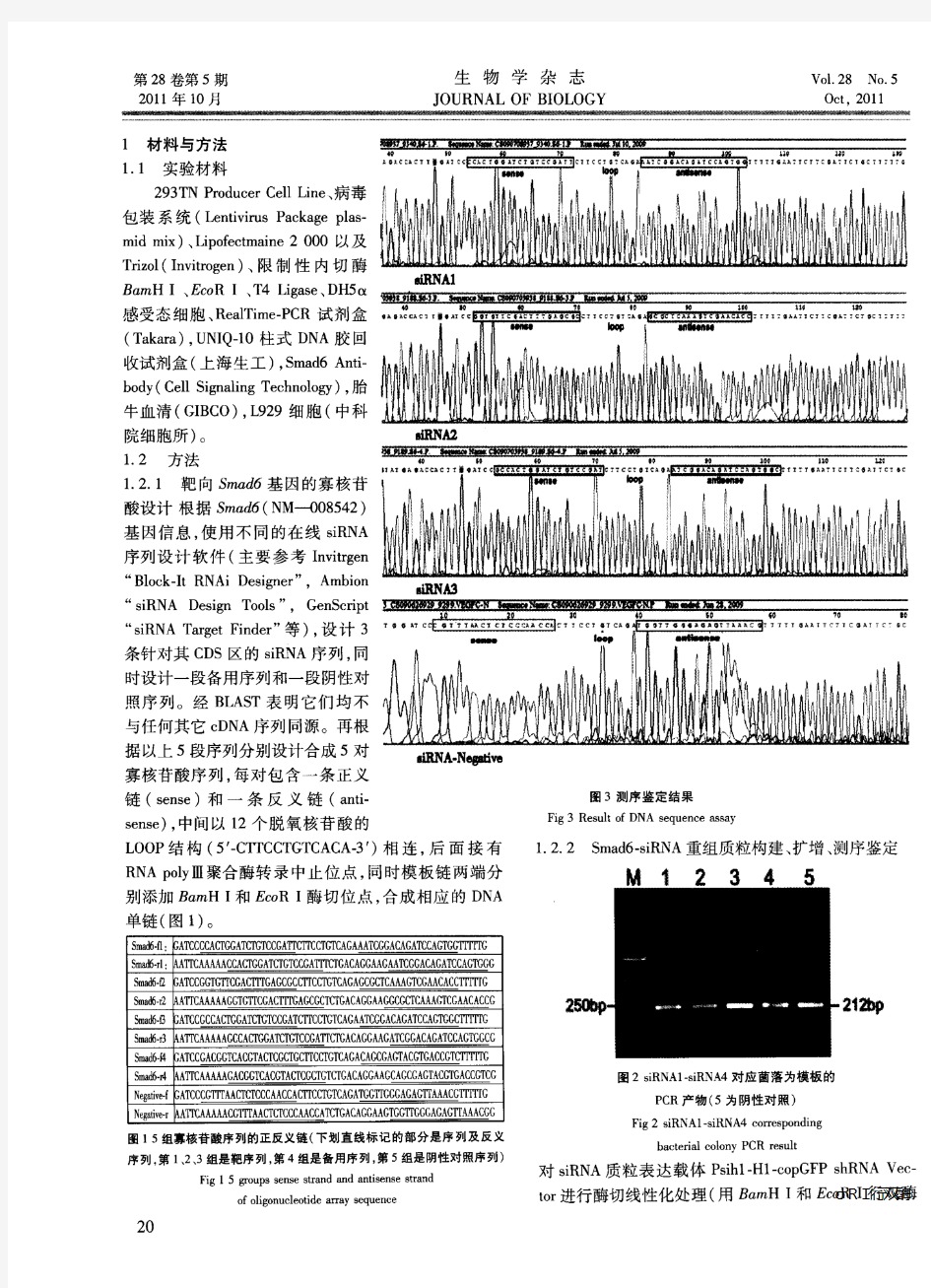
包装成分:由HIV-1基因组去除了包装、逆转录和整合所需的顺式作用序列而构建,能够反式提供产生病毒颗粒所必需的蛋白。包装成分通常被分开构建到两个质粒上,一个质粒表达Gag和Pol蛋白,另一个质粒表达Env蛋白,其目的也是降低恢复成野生型病毒的可能。将包装成分与载体成分的3个质粒共转染细胞(如人肾293T细胞),即可在细胞上清中收获只有一次性感染能力而无复制能力的、携带目的基因的HIV-1载体颗粒。 载体成分:与包装成分互补,即含有包装、逆转录和整合所需的HIV顺式作用序列,同时具有异源启动子控制下的多克隆位点及在此位点插入的目的基因。 为降低两种成分同源重组恢复成野生型病毒的可能,需尽量减少二者的同源性,如将包装成分上5′LTR换成巨细胞病毒(CMV)立即早期启动子、3′LTR换成SV40 polyA等。 一、实验流程(1和2为并列步骤) 1.慢病毒过表达质粒载体的构建 设计上下游特异性扩增引物,同时引入酶切位点,PCR(采用高保真KOD酶,3K内突变率为0%)从模板中(CDNA质粒或者文库)调取目的基因CDS区(coding sequence)连入T载体。将CDS区从T载体上切下,装入慢病毒过表达质粒载体。 2.慢病毒干扰质粒载体的构建 合成siRNA对应的DNA颈环结构,退火后连入慢病毒干扰质粒载体 3. 慢病毒载体的包装与浓缩纯化 制备慢病毒穿梭质粒及其辅助包装原件载体质粒,三种质粒载体分别进行高纯度无内毒素抽提,共转染293T细胞,转染后6 h 更换为完全培养基,培养24和48h后,分别收集富含
转基因小鼠的应用及其制 备法 学院:动物科技学院 专业班级: 学生姓名: 学号: 指导老师: 时间:2015年12月17日
老师,这篇文章是在图书馆《分子生物技术——重组DNA的原理与应用》书上整理下来的,没有参考文献,但是,这一万多字都是自己亲手打下来的,图也是自己用软件画的,其中的原理也都 弄懂,愿老师见谅。 转基因小鼠的应用及其制备法
(西北农林科技大学动物科技学院,凌712100) 摘要随着后基因组时代的到来,转基因动物已成为新兴的最有效的实验模型。从上世纪80年代以来,上百个不同的基因已经转入各种品系的小鼠中。这有助于理解基因调控,肿瘤发展,免疫特异性,发育分子遗传学以及人们感兴趣的其他生物学过程。转基因小鼠也已在探索利用家畜进行人类治疗药物工业化生产的可能性,以及建立各种人类遗传病的转基因生物医学模型中起到重要作用。现就制备转基因小鼠的实验法及应用前景作简单介绍。 关键词医学模型;试验法;应用前景 Methods and Applications of Transgenic Mice (Northwest A&F University,Colledge of Animal Science and Technoledge,Yangling, Shaanxi,712100,China ) Abstact:Following arrival of the post-genomics era,transgenic animals have become the most effective novel experimental model.Since the 1980’s.Hundreds of different genes have been transferred to the various strains of mice.This helps to understand the gene regulation,tumor development,immune specificity,developmental molecular genetics and other biological processes which people are interested in.Transgenic mice have also been exploring the possibility of using domestic animals for the industrial production of drugs for human treatment,and they also play an important role in establishment of a variety of genetically modified biomedical model of human genetic diseases. The article here is an overview of experimental methods and the application prospect of transgenic mice. Key words:biomedical model; experimental methods; application prospect 1990年人类基因组计划正式启动,经过13年的努力,人类基因组序列图绘制成功及人类基因组计划的所有目标全部实现。人们迎来一个崭新的时代——后基因组时代,即在基因组静态的碱基序列逐步清楚之后而最基因组进行动态的生物学功能研究。转基因动物在后基因组时代已成为生命科学研究中新 作者简介:本科,动物科学专业。Email:lw5166@126.;Tel: *通讯作者:E-mail:mailto:zhangjianqin1356@126.
课题-基因敲除小鼠的pcr鉴定
一、技术介绍与研究进展 转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,至今已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,在小鼠模型构建方面日趋完善,并且如同剪切酶和抗体等常规分子生物学试剂的制备技术一样,逐渐从基础研究实验室转向商业模式,成为一项高度标准化的新兴产业,催生了数以百计的创新药物和数以千计的优秀文章。尽管如此,传统技术仍然存在一些难以克服的缺陷,如步骤繁琐、周期漫长、成功率低、费用高昂等,而ZFN和TALEN 等新技术的出现,或有可能将这一局面彻底改变。 二、同源重组技术原理 基因敲除鼠技术是上世纪80年代中后期基于DNA同源重组的原理发展起来的,Capecchi和Smithies在1987年根据同源重组(homologous recombination)的原理,首次实现了ES的外源基因的定点整合(targeted integration),这一技术称为"基因打靶"(gene targeting)或"基因敲除"
(gene knockout),利用这种ES的显微注射就可以制作出基因敲出小鼠(KO Mice: knockout mice);由于这一工作,Capecchi和Smithies于2007年与Evans分享了诺贝尔医学奖。 同源重组(homologous recombination)定义:是指发生在姐妹染色单体(sister chromatin) 之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。在基因敲除小鼠制作过程中,需要针对目的基因两端特异性片段设计带有相同片段的重组载体,将重组载体导入到胚胎干细胞后外源的重组载体与胚胎干细胞中相同的片段会发生同源重组,如图1所示: 图1.基因敲除鼠制作同源重组原理示意图 三、制作流程
慢病毒载体,稳定表达 一、慢病毒 逆转录病毒(Retrovirus):是一种RNA病毒,在复制时需在逆转录酶的作用下首先将RNA 转变为cDNA,再在DNA复制、转录、翻译等蛋白酶作用下扩增。主要包括RNA肿瘤病毒、慢病毒及泡沫病毒等三种亚科。 慢病毒(Lentivirus):属于逆转录病毒科,名称源自该种病毒长达数年的潜伏期。 最经典的慢病毒是由HIV病毒改造而来,而且HIV-1/HIV-2系统也得到了广泛的应用,除了HIV病毒系统以外,后续还有猿类免疫缺陷病毒(simian immunodeficiency virus, SIV)载体系统、猫免疫缺陷病毒(felines immunodeficiency virus, FIV)载体系统、绵羊梅迪-维斯纳病毒(MMV)载体系统和马传染性贫血(EIA)载体系统等。 慢病毒结构: 2个调节基因: (1)tat基因:反式激活因子,对HIV基因起正调控作用。 (2)rev基因:病毒蛋白表达调节因子,增加gag和env基因对结构蛋白的表达。 4个辅助蛋白(附属)基因: (1)vif和vpu调节感染性病毒颗粒的产生; (2)vpr和nef参与疾病的表现。 慢病毒的优势: 1.慢病毒携带的基因组可整合到宿主基因组,使宿主细胞长时间稳定表达外源基因; 2.可感染分裂和非分裂细胞; 3.低免疫原性,直接注射活体组织不易造成免疫反应,适用于动物实验; 4.可以更换特异性启动子; 5.野生型的HIV大小约为9.8 kb,插入片段可长达5-6 kb;
二、慢病毒载体 慢病毒载体(Lentivirus)是一类改造自人免疫缺陷病毒(HIV)的病毒载体,是逆转录病毒的一种,基因组是RNA,其毒性基因已经被剔除并被外源性目的基因所取代,属于假型病毒。可利用逆转录酶将外源基因整合到基因组中实现稳定表达,具有感染分裂期与非分裂期细胞的特性。 慢病毒包装过程: 慢病毒基因组进入细胞后,在细胞浆中反转录为DNA,形成DNA整合前复合体,进入细胞核后,DNA整合到细胞基因组中。整合后的DNA转录成mRNA,回到细胞浆中,表达目的蛋白;或产生小RNA。慢病毒介导的基因表达或小RNA干扰作用持续且稳定,并随细胞基因组的分裂而分裂。 慢病毒包装和侵染细胞的过程(元和生物) 三、慢病毒的使用和优势 慢病毒的使用量的取决因素:滴度,感染体积,MOI ,细胞密度 滴度(Titer):单位体积液体中有感染能力的病毒或噬菌体数目。单位:TU/mL (活性滴度单位)、copies/mL (物理滴度单位) 检测方法:定量PCR检测干扰后细胞基因组中外源DNA拷贝数。 实验原理:慢病毒介导外源基因以逆转录方式整合进目的细胞基因组。 图3 MOI(multiplicity of infection):感染复数或者复感染指数。指感染时病毒和细胞数量的比值。在实验中也将某个细胞达到80%感染时所需的MOI 值定义为这个细胞的MOI值。加的病毒量(μl)=细胞数×MOI/滴度(…/ml) ×1000。 最后,818 一些有关慢病毒方面的产品: 1.关于慢病毒载体构建方面: ORF表达克隆产品【LPP-货号-载体-100,ORF/Promoter/lncRNA慢病毒】 shRNA克隆产品【LPP-货号-载体-050,shRNA慢病毒】 miRNA克隆产品【LPP-货号-载体-050,miRNA/inhibitor慢病毒】
碱法提取小鼠总DNA及基因鉴定: 一、实验器材:加样枪(1ml、200ul、20ul、10ul)、枪头(大中小一套)、EP管(20ul、1.5ml、 10ml)、试管架、浮标、温度计、胶布、手套、记号笔、锥形瓶、称量匙、冰盒 二、实验试剂:A液、B液、引物、mix、双蒸水、三蒸水、琼脂糖、TBE(5x、1x、回收液)、 核苷酸染料、Marker 三、母液配置: 1.10M NaOH:NaOH 40g加双蒸水至90ml,待NaOH完全溶解冷却后定容至100ml 2.0.5M EDTA:EDTA.Na2盐18.61g, NaOH 1.5g, 加双蒸水至80ml,逐滴加入10M NaOH 至EDTA完全溶解后加双蒸水定容至100ml 3.1M TrisHCl(pH8.0):Tris碱12.1g,加水至70ml,边搅拌边加入浓盐酸4ml,然后 边逐滴加入1M HCl边测PH值,直至PH升至8.0(+\-0.05),定容至100ml(pH=8.8 的TrisHCl中边加入浓盐酸边测PH值至PH=8.0(+\-0.05)为止) 四、实验步骤: 1.剪取鼠尾(约芝麻大小)储存于-20度冰箱(-20度冰箱,主要是防止DNA降解,4度不 行) 2.提取DNA: 1)配置工作液——20ml体系液:50ul 10M NaOH 加双蒸水至20ml 8ul 0.5M EDTA B液:800ul 1M TrisHCl(pH8.0)加双蒸水至20ml 2)加150ulA液(液体应完全浸没标本),95度水浴锅煮1.5h(将EP管插入浮标 中后用胶布缠好防止EP管在加热过程中爆开) 3)加150ulB液,混匀(上下颠倒3-5下) 4)12000r/min 4度离心5分钟(可储存于-20度冰箱) 3.PCR(冰上操作):P1 0.5ul(P为AC3I,G为AAA) 1)配置PCR体系——15ul体系P2 0.5ul Mix 7.5ul 三蒸水4.5ul DNA 2ul 2)加2ul上述离心后的上清液至PCR体系,瞬离 3)PCR仪扩增,参数设定:预变性:94度——3min 变性:94度——30s 退火:55度——30s 30个循环 延伸:72度——24s 72度——5min 4度——∞ 4.琼脂糖凝胶电泳: 1)制胶——2%琼脂糖凝胶配方: 总体积(ml)20 30 40 50 60 120 琼脂糖(g)0.4 0.6 0.8 1 1.2 2.4 TBE 1x(ml)20 30 40 50 60 120 Tris-base 13.6g TBE 5x配方:硼酸 6.56g EDTA 0.73g
基因敲除小鼠的PCR鉴定 一、实验目的: 通过PCR扩增程序及琼脂糖凝胶电泳方法鉴定凝血因子IX基因敲除小鼠的基因型。 二、实验原理: 真核生物的一切有核细胞(包括培养细胞)都能用来制备基因DNA。真核生物的DNA是以染色体的形式存在于细胞核内,因此,制备DNA的原则是既要将DNA与蛋白质、脂类和糖类等分离,又要保持DNA分子的完整。提取DNA的一般过程是将分散好的组织细胞在含SDS(十二烷基硫酸钠)和蛋白酶K的溶液中消化分解蛋白质,再用酚和氯仿/异戊醇抽提分离蛋白质,得到的DNA溶液经乙醇沉淀使DNA从溶液中析出。 1.PCR原理: PCR技术的基本原理类似于DNA的天然复制过程,其特异性依赖于与靶序列两端互补的寡核苷酸引物。PCR由变性-退火-延伸三个基本反应步骤构成: 1) 模板DNA的变性:模板DNA经加热至93℃左右一定时间后,使模板DNA双链或经PCR扩增形成的双链DNA解离,使之成为单链,以便它与引物结合,为下轮反应作准备; 2) 模板DNA与引物的退火(复性):模板DNA经加热变性成单链后,温度降至55℃左右,引物与模板DNA单链的互补序列配对结合; 3) 引物的延伸:DNA模板-引物结合物在TaqDNA聚合酶的作用下,以dNTP为反应原料,靶序列为模板,按碱基配对与半保留复制原理,合成一条新的与模板DNA 链互补的半保留复制链重复循环变性-退火-延伸三过程,就可获得更多的“半保留复制链”,而且这种新链又可成为下次循环的模板。每完成一个循环需2~4分钟,2~3小时就能将待扩目的基因扩增放大几百万倍 2.琼脂糖凝胶电泳原理: 在pH8.0~8.3的缓冲液中,核酸分子带负电荷,向正极移动。由于不同大小和构象的核酸分子电荷密度大致相同,因此在自由泳动时,各种核酸分子的迁移率相似,无法分开。然而,在浓度适当的凝胶中,由于分子筛效应,使大小和构象不同的核酸迁移率出现差异,从而把它们分开。核酸在凝胶中的迁移率取决于其分子大小、高级结构、胶浓度和电场强度,与分子的碱基组成及电泳温度(4~30℃之间)无明显关系。一般说,同样构象的分子迁移率与分子量对数及胶浓度成反比,与电场强度(小于5V/cm)成正比。 三、实验操作 1.获取鼠尾组织 剪鼠尾0.5cm置于试管中,加入500ul裂解液和10ul蛋白酶K(20mg/ml),55℃水浴过夜,至鼠尾溶解 2.提取基因组DNA 1) 试管中加入300ul饱和NaCl,充分混匀,12500rpm 离心20min
一、技术介绍与研究进展 转基因、基因敲入/敲除动物技术已经成为现代生命科学基础研究和药物研发领域不可或缺的重要技术,该技术从上世纪七八十年代诞生以来,至今已有近四十年的历史,经典技术如DNA原核显微注射、胚胎干细胞显微注射技术一直以来经久不衰,在小鼠模型构建方面日趋完善,并且如同剪切酶和抗体等常规分子生物学试剂的制备技术一样,逐渐从基础研究实验室转向商业模式,成为一项高度标准化的新兴产业,催生了数以百计的创新药物和数以千计的优秀文章。尽管如此,传统技术仍然存在一些难以克服的缺陷,如步骤繁琐、周期漫长、成功率低、费用高昂等,而ZFN和TALEN 等新技术的出现,或有可能将这一局面彻底改变。 二、同源重组技术原理 基因敲除鼠技术是上世纪80年代中后期基于DNA同源重组的原理发展起来的,Capecchi和Smithies在1987年根据同源重组(homologous recombination)的原理,首次实现了ES的外源基因的定点整合(targeted integration),这一技术称为"基因打靶"(gene targeting)或"基因敲除"(gene knockout),
利用这种ES的显微注射就可以制作出基因敲出小鼠(KO Mice: knockout mice);由于这一工作,Capecchi和Smithies于2007年与Evans分享了诺贝尔医学奖。 同源重组(homologous recombination)定义:是指发生在姐妹染色单体(sister chromatin) 之间或同一染色体上含有同源序列的DNA分子之间或分子之内的重新组合。在基因敲除小鼠制作过程中,需要针对目的基因两端特异性片段设计带有相同片段的重组载体,将重组载体导入到胚胎干细胞后外源的重组载体与胚胎干细胞中相同的片段会发生同源重组,如图1所示: 图1.基因敲除鼠制作同源重组原理示意图 三、制作流程
慢病毒包装系统简介及应用 一、慢病毒包装简介及其用途 慢病毒(Lentivirus )载体是以HIV-1 (人类免疫缺陷I 型病毒)为基础发展起来的基因治疗载体。区别一般的逆转录病毒载体,它对分裂细胞和非分裂细胞均具有感染能力。慢病毒载体的研究发展得很快,研究的也非常深入。该载体可以将外源基因有效地整合到宿主染色体上,从而达到持久性表达。在感染能力方面可有效地感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞、干细胞等多种类型的细胞,从而达到良好的的基因治疗效果,在美国已经开展了临床研究,效果非常理想,因此具有广阔的应用前景。 目前慢病毒也被广泛地应用于表达RNAi 的研究中。由于有些类型细胞脂质体转染效果差,转移到细胞内的siRNA 半衰期短,体外合成siRNA 对基因表达的抑制作用通常是短暂的,因而使其应用受到较大的限制。采用事先在体外构建能够表达siRNA 的载体,然后转移到细胞内转录siRNA 的策略,不但使脂质体有效转染的细胞种类增加,而且对基因表达抑制效果也不逊色于体外合成siRNA ,在长期稳定表达载体的细胞中,甚至可以发挥长期阻断基因表达的作用。在所构建的siRNA 表达载体中,是由RNA 聚合酶Ⅲ启动子来指导RNA 合成的,这是因为RNA 聚合酶Ⅲ有明确的起始和终止序列,而且合成的RNA 不会带poly A 尾。当RNA 聚合酶Ⅲ遇到连续4 个或5 个T 时,它指导的转录就会停止,在转录产物3' 端形成1~4 个U 。U6 和H1 RNA 启动子是两种RNA 聚合酶Ⅲ依赖的启动子,其特点是启动子自身元素均位于转录区的上游,适合于表达~21ntRNA 和~50ntRNA 茎环结构(stem loop )。在siRNA 表达载体中,构成siRNA 的正义与反义链,可由各自的启动子分别转录,然后两条链互补结合形成siRNA ;也可由载体直接表达小发卡状RNA(small hairpin RNA, shRNA),载体包含位于RNA 聚合酶Ⅲ启动子和4 ~5 T转录终止位点之间的茎环结构序列,转录后即可折叠成具有1~4 个U 3 ' 突出端的茎环结构,在细胞内进一步加工成siRNA 。构建载体前通常要通过合成siRNA 的方法,寻找高效的siRNA ,然后从中挑选符合载体要求的序列,将其引入siRNA 表达载体。 慢病毒载体(Lentiviral vector )较逆转录病毒载体有更广的宿主范围,慢病毒能够有效感染非周期性和有丝分裂后的细胞。慢病毒载体能够产生表达shRNA 的高滴度的慢病毒,在周期性和非周期性细胞、干细胞、受精卵以及分化的后代细胞中表达shRNA ,实现在多种类型的细胞和转基因小鼠中特异而稳定的基因表达的功能性沉默,为在原代的人和动物细胞组织中快速而高效地研究基因功能,以及产生特定基因表达降低的动物提供了可能性。 慢病毒表达载体包含了包装、转染、稳定整合所需要的遗传信息。慢病毒包装质粒可提供所有的转录并包装RNA 到重组的假病毒载体所需要的所有辅助蛋白。为产生高滴度的病毒颗粒,需要利用表达载体和包装质粒同时共转染细胞,在细胞中进行病毒的包装,包装好的假病毒颗粒分泌到细胞外的培养基中,离心取得上清液后,可以直接用于宿主细胞的感染,目的基因进入到宿主细胞之后,经过反转录,整合到基因组,从而高水平的表达效应分子。 二、这一系统的目的,主要是为了解决以下问题: 1. 对于一些较难转染的细胞,如原代细胞、干细胞、不分化的细胞等,能大大提高目的基因转导效率,而且目的基因整合到宿主细胞基因组的几率大大增加,这就为RNAi,cDNA 克隆以及报告基因的研究提供了一个有利的途径。 2. 进行稳转细胞株的筛选; 3. 为活体动物模型实验提供高质量的包含目的基因的病毒液; 在细胞相关的实验操作中,对于一些按常规方法难以转染甚至无法转染的细胞,通过病毒介导的实验能够大大提高基因的转导效率,以达到目的基因的高效瞬时表达。
PCR方法在ApoE基因敲除小鼠基因型鉴定中的应用 摘要目的为ApoE基因敲除鼠探索快速、简单的基因型PCR检测方法。方法设计两对引物扩增野生型ApoE基因和ApoE缺陷突变基因的DNA片段,用PCR仪梯度方案测试最佳退火温度,然后用PCR鉴定方案检测小鼠基因型并将所得基因型结果与已经过经典的Southern blot方法检测得到的基因型结果比较。结果野生型仅在155 bp处有一条条带,突变纯舍子仅在245 bp处有一条条带,杂合子则在155和245bp处出现两条条带。用PCR方法获得的ApoE基因分析结果与经典的Southern blot方法获得的结果完全一致。结论用PCR方法分析ApoE基因敲除鼠的基因型具有快速、简单、廉价和适用的特点。 关键词聚合酶链反应基因型基因打靶载脂蛋白E Genotype identification of ApoE Gene Knockout Mice with Polymerase Chain Reaction Objective This study was to explore a simple and quick polymerase chain reaction(PCR)method for genotyping of ApoE knockout mice.Method Two pairs of primers were designed to amplify genomic DNA fragment of wild-type ApoE gene and the same region on ApoE targeting veceor respectively.A gradient PCR strategy was used to test the best annealing temperature. Results A 155bp band was found in wild-type ApoE mice,a 245 bp band in homozygous mutated ApoE mice and both bands in hetemzygous mice.The genotyping results were completely coincided with those from typical Southern blot. Conclusion PCR is a simple,fast and practical method for the genotyping of ApoE gene knockout mice. Key words Poiymerase Chain Reaction genotype gene targeting ApoE 载脂蛋白(apolipoprotein,ApoE)是清除乳糜微粒和极低密度脂蛋白的受体的配体,因此,缺乏ApoE则会导致血液循环中富含胆同醇的物质积累而更加容易引起动脉粥样硬化