
50个全外显子测序揭示人类的高原适应机制
Xin Yi等。
Science 329, 75 (2010);
DOI: 10.1126/science.1190371
50个全外显子测序揭示人类的高原适应机制
生活于青藏高原的藏族人表现出了对极端高原环境的遗传适应性。我们对50个藏族人进行全外显子基因组测序,它们包含了92%的人类基因编码序列,人均覆盖度为18倍。基因分析显示了该特殊人群等位基因频率的变化,表明这些人对高原环境具有很强的适应性。
研究显示,表现出最强自然选择信号的基因是编码内皮细胞含PAS结构域蛋白-1(EPAS1)的基因,这是一个参与应答缺氧的转录因子。研究显示,EPAS1基因的一个单核苷酸多态性(SNP)在78%的藏族和汉族人群中存在差异,这是目前发现的速率改变最快的等位基因。该单核苷酸多态性与红细胞丰度的关联分析也支持EPAS1改变在适应缺氧环境中的作用,进一步表明它是适应高原环境的一个重要的遗传位点。
在广袤的大自然中生存的人类可能会存在文化和基因上的适应。其中人类面临的最严厉的环境挑战就是高海拔地区(如青藏高原)的低含氧量。这一地区的许多居民在海拔4000米以上居住,那里的氧气浓度比海平面大约低40%。藏族对缺氧环境有着他们自己的遗传适应性,如出生体重(1),血红蛋白水平(2),婴儿(3)和运动后的成年人(4)血液中的氧饱和度。这些结果暗示了高原适应机制的自然选择历史,我们对整个基因组的遗传差异进行分析,可能会发现这一点。
我们对中国西藏自治区海拔4300米以上(5)的两个村庄里的50个非亲个体进行全外显子基因组测序。针对将近两万个基因的外显子和侧翼区的34Mb序列,利用罗氏NimbleGen公司(威斯康星州麦迪逊市)的2.1M外显子序列捕获芯片(6)将其富集。测序采用了Illumina公司(加利福尼亚州圣地亚哥市)的基因组分析仪II平台,并使用序列比对程序SOAP(7)将测序片段比对到人类参考基因组序列上[美国生物技术信息中心(NCBI) 36. 3版]。
1深圳华大基因研究院,中国深圳,518083。
2中国科学院研究生院,中国北京,100062。
3加州大学伯克利分校综合生物学与统计系,美国加州,94820。4华南理工大学生物系本科创新班,中国广州,510641。
5西藏自治区人民医院,中国拉萨,850000。
6加州大学戴维斯分校进化与生态学系,美国加州,95616。
7哥本哈根大学生物系,丹麦哥本哈根,1165。
8华南理工大学理学院本科创新班,中国广州,510641。
9深圳大学医学院基因组研究所,中国深圳,518060。
10拉萨市人民医院,中国拉萨,850000。
11西藏军区总医院,中国拉萨,850007。
12西双版纳傣族自治州人民医院,中国云南景洪,666100。
*以上机构及相关人员对本研究作出了贡献。如有疑问请联系:
E-mail:wangjian@https://www.doczj.com/doc/25463102.html, ( Ji.W.);
wangj@https://www.doczj.com/doc/25463102.html, ( Ju.W.);
rasmus_nielsen@https://www.doczj.com/doc/25463102.html, (R.N.)
全外显子测序的平均深度为18倍(表S1),但这并不能保证个别基因型的准确性。因此,我们用贝叶斯统计法(5)估算出每个可能的基因型概率,从而估算出单核苷酸多态性(SNP)的概率和每个位点的人类等位基因频率。在藏族样本中总共151825个SNPs有超过50%被识别出是可变的,有101668个超过99%的SNP是可变的(表S2)。Sanger测序验证了56个SNPs 中的53个,至少包含95%的SNP和3%~50%的次等位基因频率。等位基因频率的估算值显示存在过量的低频变异(图S1),特别是在非同义SNPs中。
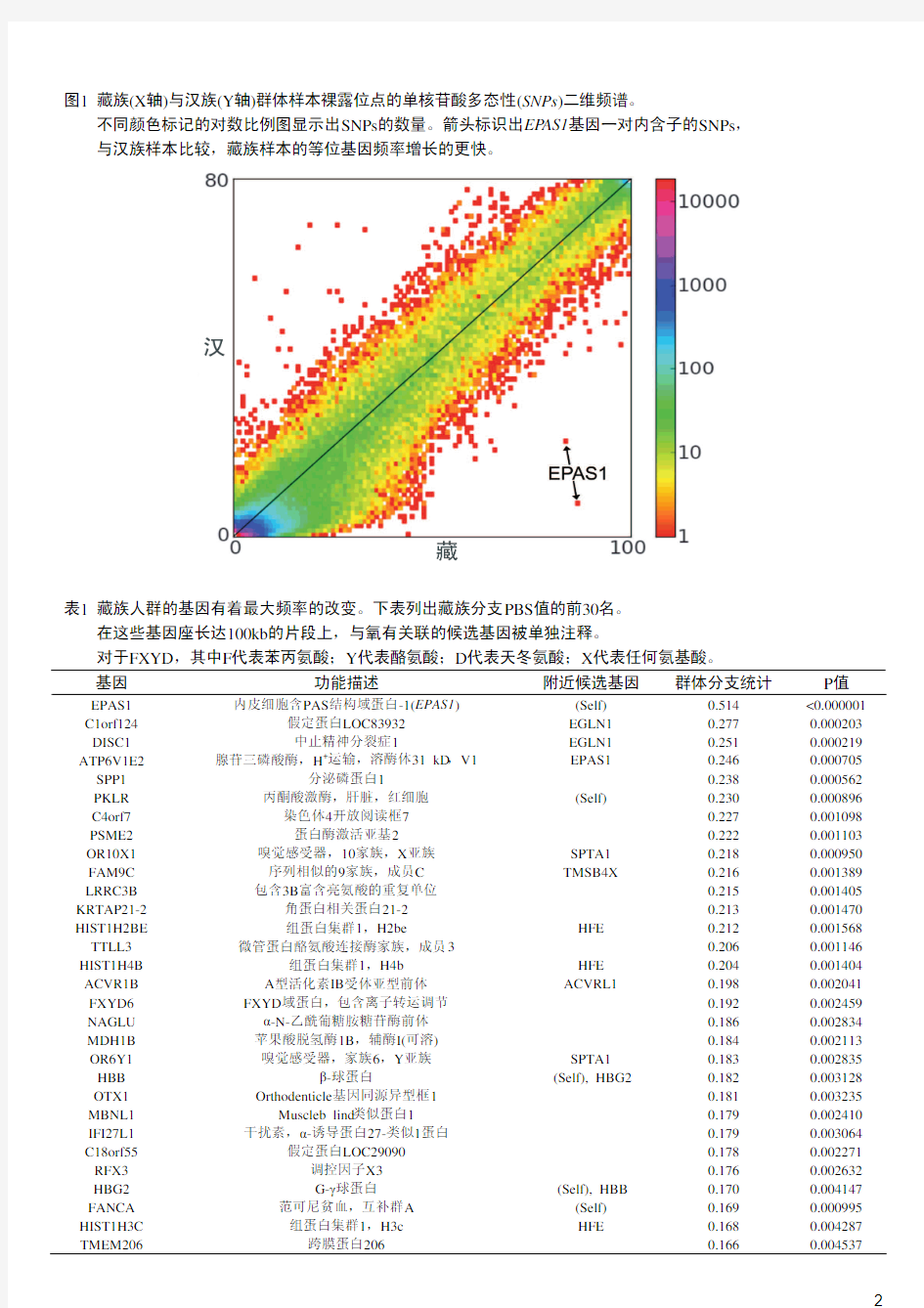
该数据与来自于北京的40个汉族人的基因组进行比较[样本来自于中测检测(CHB)人类基因组单体型图计划(HapMap),属于1000个基因组计划的一部分(https://www.doczj.com/doc/25463102.html,)],测序得出汉族人均大约4倍的覆盖度。北京的海拔不超过50米,几乎所有的汉族人来自于海拔2000米以下。在较低的遗传分化基础上汉族人样本与藏族人样本形成鲜明的对比(F ST= 0.026)。这两个藏族村庄是体现该族遗传结构的最小单位(F ST= 0.014),因此,我们可以将他们当着整个群体进行分析。我们观察到,汉藏之间的等位基因频率有着很强的协方差(图1),但是过量的SNPs在汉族中频率很低,在藏族中频率中等。
从两个群体同义位点的二维频谱,可以估算出人类历史模型(8)。最佳拟合模型表明,藏族和汉族人群在2750年前出现分化,汉族人口从最初的小规模逐渐增大,藏族人口从最初的大规模逐渐减少(图S2)。这估计是由于藏族人移民至汉族区,双方长期相互渗透所造成的。
图1 藏族(X轴)与汉族(Y轴)群体样本裸露位点的单核苷酸多态性(SNPs)二维频谱。
不同颜色标记的对数比例图显示出SNPs的数量。箭头标识出EP AS1基因一对内含子的SNPs,
与汉族样本比较,藏族样本的等位基因频率增长的更快。
表1 藏族人群的基因有着最大频率的改变。下表列出藏族分支PBS值的前30名。
在这些基因座长达100kb的片段上,与氧有关联的候选基因被单独注释。
对于FXYD,其中F代表苯丙氨酸;Y代表酪氨酸;D代表天冬氨酸;X代表任何氨基酸。
基因功能描述附近候选基因群体分支统计P值EPAS1 内皮细胞含PAS结构域蛋白-1(EPAS1) (Self) 0.514 <0.000001 C1orf124 假定蛋白LOC83932 EGLN1 0.277 0.000203 DISC1 中止精神分裂症1 EGLN1 0.251 0.000219 ATP6V1E2 腺苷三磷酸酶,H+运输,溶酶体31 kD,V1 EPAS1 0.246 0.000705 SPP1 分泌磷蛋白1 0.238 0.000562 PKLR 丙酮酸激酶,肝脏,红细胞(Self) 0.230 0.000896 C4orf7 染色体4开放阅读框7 0.227 0.001098 PSME2 蛋白酶激活亚基2 0.222 0.001103 OR10X1 嗅觉感受器,10家族,X亚族SPTA1 0.218 0.000950 FAM9C 序列相似的9家族,成员C TMSB4X 0.216 0.001389 LRRC3B 包含3B富含亮氨酸的重复单位0.215 0.001405 KRTAP21-2 角蛋白相关蛋白21-2 0.213 0.001470 HIST1H2BE 组蛋白集群1,H2be HFE 0.212 0.001568 TTLL3 微管蛋白酪氨酸连接酶家族,成员3 0.206 0.001146 HIST1H4B 组蛋白集群1,H4b HFE 0.204 0.001404 ACVR1B A型活化素IB受体亚型前体ACVRL1 0.198 0.002041 FXYD6 FXYD域蛋白,包含离子转运调节0.192 0.002459 NAGLU α-N-乙酰葡糖胺糖苷酶前体0.186 0.002834 MDH1B 苹果酸脱氢酶1B,辅酶I(可溶) 0.184 0.002113 OR6Y1 嗅觉感受器,家族6,Y亚族SPTA1 0.183 0.002835 HBB β-球蛋白(Self), HBG2 0.182 0.003128 OTX1 Orthodenticle基因同源异型框1 0.181 0.003235 MBNL1 Muscleb lind类似蛋白1 0.179 0.002410 IFI27L1 干扰素,α-诱导蛋白27-类似1蛋白0.179 0.003064 C18orf55 假定蛋白LOC29090 0.178 0.002271 RFX3 调控因子X3 0.176 0.002632 HBG2 G-γ球蛋白(Self), HBB 0.170 0.004147 FANCA 范可尼贫血,互补群A (Self) 0.169 0.000995 HIST1H3C 组蛋白集群1,H3c HFE 0.168 0.004287 TMEM206 跨膜蛋白206 0.166 0.004537
在群体间,拥有较强频率差异的基因是自然选择的潜在目标。然而,简单的F ST值排序并不能表明哪个群体被自然选择所影响。所以,我们通过加入与之有较远亲缘关系的第三群体,来估算特异人群等位基因频率的变化。因此,我们检测了200个丹麦人的全外显子序列,收集并分析,用来对藏族样本进行描述。通过比较这三个群体样本的三对F ST值,我们可以估算出从汉族人群分化出来以后,藏族人群频率的变化(5,9)。我们发现,群体分支统计(PBS)在探究最近发生的自然选择上起着重要的作用(图S3)。
藏族人基因的极端PBS值显示出他们对高原环境具有很强的遗传适应性,最强的这些信号包含若干基因,已知它们具有输送氧气和调节作用(表1和表S3)。总的来说,我们数据集里的34个基因(可归入基因本体论的“缺氧应答”类别)比全基因组的平均值具有更加显著的PBS 值(P=0.00796)。
表现出最强自然选择信号的基因是编码内皮细胞含PAS结构域蛋白-1(EPAS1)的基因。基于丹麦人,汉族人和藏族人之间的频率差异,相对于其他基因,EP AS1基因在藏族分支中的存在时间更长(图2)。为了证明自然选择的作用,我们将人口预测模型的中性模拟值与PBS值进行比较。观察EP AS1发现,一百万次模拟没有一次超过PBS值。在校正了测试基因的数目后,这一结果仍然是显著的(Bonferroni校正后,P<0.02)。许多其他未经校正的基因,P值小于0.005(表1),虽然多个测试在校正后没有一个是统计显著的,但是一些具有很多功能的基因表明,它们可能也有助于人类对高原环境的适应。
EP AS1也被称为缺氧诱导因子2α (HIF-2α)。HIF家族的转录因子由两个亚基组成,有三个候补α亚基(HIF-1α, HIF-2α/EP AS1, HIF-3α),二聚体的αβ亚基由ARNT(芳香羟受体核易位子) 或ARNT2编码。HIF-1α和EP AS1都有自己独特的调控目标(10),EP AS1狭义的表达型包括成人和婴儿的肺、胎盘和血管内皮细胞(11)。EP AS1的一个蛋白质稳定突变与红细胞增多有关(12),这表明EP AS1与红细胞产生的调控有关。
尽管我们的测序主要是针对外显子,但是一些侧翼区的内含子和非编码区(UTR)也被列入其中。汉族和藏族人的EP AS1内含子的SNP有最大的频率差异(在汉族人样本中的等位基因频率为9%,藏族中为87%,表S4),而没有氨基酸变异在人群中的频率超过6%。直接导致变异体产生的可能是自然选择作用,或其他有关系的非编码变体影响EP AS1调控的结果。详细的分子研究就需要调查,与基因表达的变化方向和幅度有关联的SNP,对组织和发育时间点的影响,以及下游靶基因对调控的改变。
已经证明EP AS1的SNPs与健壮的体质有关联(13)。我们的数据集包含一套不同的SNPs,我们对有最极端频率差异的SNP进行了关联性检测,刚好位于上游的第6个外显子上。对此SNP进行等位基因与血液表型关联性的检测,没有显示出与氧饱和度有关。然而,发现红细胞数(F test P=0.00141)与血红蛋白浓度(F test P=0.00131)有明显的关联,当对每个村分别进行检测时,这两个性状有显著或轻微显著的P值(表S5)。将48个不相关的SNPs与EPAS1的SNP基因型数据进行比较,证实他们的P值是很极端的(图S4)。
西藏群体样品中高频率的等位基因是与少量的红细胞和含量相对较低的血红蛋白相联系的(表S4)。产生较多的红细胞是应对缺氧的正常应激反应。这可能就是藏族EP AS1等位基因的携带者在高海拔地区能够维持氧供应,而没有必要提高红细胞水平的原因。所以,这里观察到的血红蛋白差异并不能解释选择的目的表型,反而得出的结果可能是EP AS1的介导对缺氧环境具有副作用。虽然尚未发现确切的生理学机制,但结果表明,通过对等位基因有针对性的选择,很可能赋予其适应高原缺氧环境的相关功能。
我们还检测了在近似于自然选择下的成人和婴儿的血红蛋白组分(各自的HBB和HBG2)。这些基因位点相距仅20kb(图S5),所以他们的PBS值可以反映出一个自适应事件。对于这两个基因,藏族与汉族SNP频率的内含子部分差异最大。虽然已经在一些高原适应物种中发现了变异球蛋白(14),但似乎更有可能变异由这些基因所调控。有人报道了安第斯高地的一个类似情况,HGB2启动子的变异随着海拔的变化而变化,并且与胎儿到成人血红蛋白的滞后转换相关(15)。
除了HBB外,另外两个与贫血有关的基因也被确定:F ANCA和PKLR,分别与红细胞的生产和维护相关(16,17)。我们还确定了在怀孕或分娩期由缺氧所引起的疾病的基因:精神分裂症(DISC1和FXYD6)(18,19)和癫痫症(OTX1)(20)。然而,影响DISC1与C1orf124选择的强信号可能要追溯到EGLN1的调控区,该调控区位于这些位点(图S5)与缺氧反应途径的功能区域之间(21)。
在这项研究中发现的其他基因也位于候选基因附近。OR10X1和OR6Y1在SPTA1内部大约60kb内(图S5),它与红细胞的形态有关(22)。此外,这项研究中的三个组蛋白(表1)都聚集在HFE周围(图S5),HFE是一个与铁的储存有关的基因(23)。群体遗传信号对邻近基因的的影响与缺氧环境所引起的近的较强的选择是一致的。如果目标候选基因调控区的适应性突变不在常见的外显子多态性附近,我们可以预测侧翼基因将会发生大的频率变化。
已经查明的这些基因中,只有EGLN1在最近的一个关于安第斯高地人SNP变化的研究中被提及(24)。这一结果与所观察到的藏族人和安第斯人的生理差异是一致的(25),表明这两种人在对高海拔的适应性进化上采取了不同的路径。
先前对喜马拉雅山居民若干基因位点的研究显示,在我们的数据集中没有任何选择的迹象(表S6),EP AS1在以前的高原研究中一直未被重视。然而EP AS1可能在
氧气的调控过程中发挥着重要作用,该基因是在对一个非候选基因组进行自然选择作用调查时被发现,进化推理后,阐明有着重要功能的基因位点的作用。
我们的估计,汉族人和藏族人在2750年以前开始分离,随后开始迁移。看来我们所关注的EP AS1的SNP可能比北欧国家的乳糖酶持久等位基因具有更快的频率改变速度,这个频率的增大经历了7500年(26)。EP AS1可能是自然选择作用于人群强有力的实证,这个基因的变异似乎对西藏地区人类的生存和(或)繁殖起着重要作用。
图2 特异群体等位基因频率变化。
(A) 依照每个基因可变化位点的数量,以藏族分支PBS统计值为基础得到的F ST分布值,红色的是边缘基因。
(B) 关于EPAS1基因的选择信号:
藏族、汉族及丹麦人的分支长度代表遗传的F ST平均值(左边);
EPAS1基因的分支长度表明沿着藏族血统出现的大量分化(右边)。
本研究由深圳华大基因研究院发起和主导,得到了国家自然科学基金委员会、国家科技部、中国科学院、深圳市政府以及丹麦、美国、瑞士等国自然科学基金委员会的支持。
背景资料(1)
深圳华大基因研究院发现青藏高原世居藏族人群高原适应的关键基因,有关这一科研成果的论文《50个全外显子测序揭示人类的高原适应机制》在最新一期美国权威学术刊物《科学》(Science)上正式发表。该成果由深圳华大基因研究院发起和主导,得到了国家自然科学基金委员会、国家科技部、中国科学院、深圳市政府以及丹麦、美国、瑞士等国自然科学基金委员会的支持。
这一成果揭示了青藏高原世居藏族人群高原适应的分子机制之谜,对预测、预防与治疗高原缺氧性疾病,促进我国高原地区社会和经济发展具有重大意义;该成果阐明了人类的基因组在极端环境下发生了何种适应性变化,具有改写人类分子进化教科书的意义。
青藏高原是世界上海拔最高、面积最大的高原,素有“世界屋脊”之称,极高海拔也使得青藏高原具有独特的地理气候环境、人文生活习惯和医疗卫生事业状况。高原环境对人体关键的影响因素是低压性低氧,大气压力随海拔增高而降低,氧分压也随之下降。当生活在低海拔地区的人来到高原环境时,由于氧分压的降低,会使人产生缺氧,因而引起“高原反应”,严重的“高原反应”会引起肺水肿和脑水肿,威胁到人的生命。而世居高原的人群在这样的环境下没有“高原反应”,将世居高原人群与低海拔人群的基因组进行对比分析,具有重要的启发意义。
藏族人群是世界上居住高原时间最长,并对高原低氧环境适应能力最佳的民族,本研究使用比较基因组学的方法阐明了高原世居藏族人群的低氧适应机制。“应用先进的基因组学分析技术——全外显子测序技术,对青藏高原世居藏族人群和低海拔人群进行比较,我们发现了藏族人群适应高原环境的关键基因。”该研究负责人、深圳华大基因研究院汪建研究员说。利用第二代高通量测序技术对50个藏族人的全基因组外显子进行测序,并将结果与低海拔汉族人群以及高加索人群的外显子进行对比,通过一套新开发的寻找自然选择信号的算法,计算出在藏族人群中受到自然选择的基因。这些受到自然选择的基因,就可能是在藏族人群高原适应中起着重要作用的基因。结果显示,有一系列基因在藏族人群的高原适应中发挥作用,其中EPAS1 基因可能起着关键作用。进一步通过对藏族人群中EPAS1基因的改变位点进行关联分析,发现EPAS1基因中受选择的基因型与藏族人群血红蛋白的代谢有关。EPAS1基因是HIF通路(低氧诱导调节通路)中的重要基因,在人体面对低氧环境的调节通路中起到核心作用。藏族人群特有的“EPAS1”基因不同于汉族人群,正是这种遗传基因阻止了藏族人血红蛋白浓度的过度升高,降低了各种高原性疾病发生的可能性。由于EPAS1基因与缺氧及血红蛋白生成密切相关,对这一基因的研究还有可能对某些血液性疾病的治疗带来突破,并且还可应用于运动员的筛选等方面。
同时,该成果发现了其它一些重要的高原适应相关基因,例如EGLN1基因、FANCA基因等共30个重要候选基因。这些基因可能在藏族人群的高原适应机制中发挥重要的作用,但是其明确的生理生化表型仍不是很确定,这为科学家们下一步对高原缺氧性疾病的研究指明了方向。这是我国在高原医学研究中的重要基础性突破,必将带动相关的基础研究和应用研究的发展。
背景资料(2)
由深圳华大基因研究院与美国加州大学伯克利分校、丹麦哥本哈根大学等单位合作的研究成果“对200个人类外显子的测序揭示大量低频率非同义突变的存在”在国际著名学术杂志《自然—遗传学》(Nature Genetics)上发表,这是华大基因在人类基因组研究领域取得的又一项重要成果。
该项研究对200个丹麦个体蛋白质编码基因的外显子组进行了深度测序,发现了大量以往未知的单核苷酸多态性位点(SNP),其中大部分在人群中都以较低频率出现。该研究完成了目前在人类外显子区域规模最大、分辨率最精细的遗传图谱,并以翔实的数据证明,人群当中的低频率多态性位点富集了大量能引起蛋白质氨基酸序列改变的变异,而这类变异在人群中受到自然选择作用,可能具有影响人类健康的功能。
最近有多项科学研究指出,以往对多基因控制的复杂疾病所进行的关联分析研究尽管从理论上可行,并在实践中发现了许多疾病关联基因,但却仅能解释复杂疾病遗传性的一小部分。这一现象被称为“遗传度缺失”,是当前复杂疾病基因组研究的一个主要难题。华大基因的这一研究首次证实,影响人类健康和疾病易感性的多态性位点在人群中往往频率低,但是相关位点的个数很多。既往的复杂疾病关联分析使用的基因分型芯片仅对常见多态性位点进行测定,而无法研究低频率多态性位点,从而漏掉大量疾病关联位点,造成“遗传度缺失”。该研究不仅指出了目前主流疾病研究方法的缺陷,并颠覆性地提出疾病关联分析应充分使用测序技术而非基因分型技术,从而对改变科学家对复杂疾病的研究手段,推动人类健康与医学研究的进步具有里程碑意义。
该研究是中丹合作糖尿病关联基因及变异研究(LUCAMP)项目的一部分。LUCAMP项目旨在利用新一代测序技术对1000个内脏肥胖病人和1000名对照健康人进行外显子组测序,计划将鉴定出新的与代谢疾病相关的常见突变和稀有突变。
之前,华大基因采用外显子组测序技术对50个藏族人的外显子进行测序研究并寻找到与藏族人高原适应性密切相关的候选基因。该成果被发表在《科学》杂志上(Science. 2010 July; 329(5987): 75-78)。有理由相信,外显子测序技术将会更广泛地应用于人类遗传疾病研究,特别是在遗传诊断以及新药研发等领域发挥出越来越重要的作用。
外显子组测序
外显子组测序是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。
外显子组测序在米勒综合症的研究中首次得到成功的应用[1],随后通过外显子组测序发现了歌舞伎综合症[2]、重型颅脑畸形[3]等孟德尔疾病的新的致病基因突变。最近,应用外显子组测序发现了家族低β脂蛋白血症疾病的一个新的致病突变[4] ,该疾病是一种脂质代谢障碍性的复杂疾病。这些结果表明外显子组测序可用于寻找单基因疾病、复杂疾病(如糖尿病、肥胖症等代谢综合症)、甚至是癌症的致病基因或易感基因。
1. 技术优势
?发现外显子区的绝大部分的疾病相关变异
?可发现常见变异和频率<5%低频突变
?只对基因组的约1%测序,更加经济、高效
?在相同预算情况下,可提供更高深度测序
2. 实验流程
外显子组测序的实验流程:首先将基因组DNA随机打断成随机片段文库,文库经纯化后通过与外显子捕获系统进行杂交富集,经QC检测合格后,即可上机进行高通量的测序。华大具有高通量的第二代测序平台,包括Illumina Hiseq 2000和ABI SOLiD TM4.0测序仪,均可进行高通量的测序(图1)。
3. 外显子捕获平台介绍
华大基因目前主要采用Aglient SureSelect外显子靶向序列富集系统(生物素化RNA探针)和NimbleGen SeqCap EZ (生物素化寡DNA核酸探针)人全外显子捕获系统(图2)。这两个系统都采用液相系统进行高覆盖率的外显子区域捕获。其优化的高通量操作流程,严格的内部质量控制,已成功适用于上千样本的外显子组测序研究。NimbleGen SeqCap EZ和Agilent SureSelect human all exon这两个外显子捕获平台探针的覆盖区域的详细信息见表1
4. 信息分析流程
外显子组测序的信息分析的具体流程见图3。外显子测序后获得原始数据(raw reads),经过去污染等过滤、比对参考基因组,获得比对到基因组上的Unique mapped reads。对数据进行标准的信息分析流程,包括对SNP、InDel等进行检测、注释和统计分析。同时对数据进行质控检测,包括测序深度、覆盖度均一性等分析的检测报告。另外根据项目研究需求,我们也会为合作伙伴提供个性化分析报告。
标准信息分析
?数据产出的统计
?外显子区域测序深度的直方分布图
?外显子捕获的均一性分析
?一致性序列的获得和SNPs的检测
? SNPs的注释
?插入和缺失(Indels)的检测
?插入和缺失(Indels)的注释
个性化信息分析
?氨基酸替代的预测分析
?群体SNP的获取和等位基因频率评估
?Mendelian disorder analysis 孟德尔遗传疾病分析
?基于新一代测序技术的全基因组关联(NGS-GWAS)分析
?阳性信号的检测
到目前为止,华大已顺利完成外显子组测序样
品超过6000个,每周外显子捕获建库通量达到近800个,具有成熟的技术平台和稳定的生产质量。华大基因参与了一系列国际国内重大疾病的外显
子研究计划,是世界肿瘤基因组计划的重要成员,与国内外知名的研究所、医院、大学和制药公司
等已建立了广泛而深入的合作。2010年的5月13日,华大基因在Science上发表了通过50个人外
显子测序寻找到与藏族人高原适应性密切相关的
候选基因的文章“Sequencing of 50 Human Exomes Reveals Adaptation to High Altitude”。2010年10
月4日,由华大基因与美国加州大学、丹麦哥本
哈根大学等单位在《自然-遗传学》上合作发表“对200个人类外显子的测序揭示大量低频率非同义
突变的存在” 的研究成果。
一种基于大规模并行全基因组测
序的单核苷酸多态性检测方法一、技术领域
本发明涉及一种基于新一代大规模并行全基因组测序的单核苷酸多态性的检测方法,属基因工程技术领域。
二、背景技术
遗传突变影响着生物体表型的变异,罹患疾病的风险,对药物和环境刺激的反应。全基因组连锁分析和定点克隆技术对受单基因影响的孟德尔遗传病的研究已经取得了巨大的成功。然而,绝大多数常见疾病,如糖尿病,心血管疾病和癌症等重要的数量性状具有复杂的遗传学基础,由多个基因以及基因与环境因素之间的相互作用共同决定。连锁分析在检测对疾病的影响近于中性的遗传变异时具有极大的局限性。相对于连锁分析和定点克隆来说,全基因组关联性分析能够为疾病关联基因位点定位提供一个更好的方法。人类基因组序列的测序完成,使得数以百万计的单核苷酸多态性位点得以鉴定,构建了高密度的单倍体型基因组变异图谱。这些研究进展开创了使用大规模全基因组的单核苷酸多态性检测技术来寻找引起各种人类疾病或与之相关的基因变异的时代。二十多年以来,Sanger法测序和荧光电泳技术一直在DNA测序领域占据着主导地位,使用随机鸟枪法策略或目标区域的PCR扩增法进行了大量的SNP检测。现有dbSNP
数据库中的
大多数SNP位点都是通过这些方法鉴定的。鸟枪法测序进行SNP检测的标准方法是将测序片段与基因组参考序列进行比对,并根据碱基的质量打分来过滤掉低质量的测序错误,得到较为可信的SNP结果。使用来自二倍体样本的PCR扩增序列进行直接测序,再通过对色谱图进行分析,检测出杂合的多态性位点,也是常见的方法,主要软件有SNPdetector,novoSNP,PolyPhred以及PolyScan。
与传统的毛细管电泳法测序相比,新一代测序技术如Illumina Genome Analyzer (GA), AB SOLiD,以及Roche 454 FLX系统显着地提高了测序通量,极大地降低了成本。Illumina GA一次运行可以产生约四千万条长度为50bp的测序片段。这种超高的测序通量使得新一代的测序技术特别适合于在已知参考基因组序列的基础上进行大规模个体的重测序从而进行基因变异的研究。截至当前,使用新的测序技术已经完成了两个人的基因组测序:James Watson的个人基因组测序(Roche 454FLX)和第一个亚洲人的基因组测序(Illumina GA)。此外,国际千人基因组计划执行委员会也决定使用这种测序技术对来自全世界的1000个个体基因组进行测序,得到最详细的人类基因组变异图谱。
随着新的测序技术的发展,相应的SNP检测方法也有了很好的发展;然而,由于新测序技术产生的测序片段与以往相比有显著的差异,新的精确的SNP检测方法也亟待开发。
三、发明内容
本发明的目的是提出了一种适应于大规模平行测序法Illumina GA技术特点的构建待测基因组一致序列和检测SNP的新方法。在分析了Illumina GA 测序数据的错误特征以后,解决了由于测序过程和数据处理过程中实际问题所造成的一致序列不准确及SNP不准确等问题,从而为采用新一代测序技术进行高效、快速、准确的全基因组测序分析提供了可靠的手段。
本发明提出的一致序列构建方法和SNP检出方法,包括以下步骤:
(1) 将测序数据比对到参考基因组上
将测序数据比对到参考基因组上。。
使用短序列比对程序SOAP将Illumina GA的测序片段比对到参考基因组序列上。(https://www.doczj.com/doc/25463102.html,)
(2) 对所有唯一比对上的测序片段进行统计
通过对SOAP程序结果的文本处理,统计特定测序质量值和特定测序序列坐标下,每两种碱基之间的错配比例,将此比例作为对错配概率的估计,记录在一个四维概率矩阵里面,作为统计学模型中各项参数的基础。(3) 判别基因组上每个碱基的基因型
对于基因组上每一个位点,将比对在此位点上所有的测序片段碱基收集起来,记录其碱基类型、测序质量和在测序片段上的序列坐标,从四维概率矩阵中查出四种碱基观察到测序碱基的概率。对于二倍体基因组而言,其真实基因型的可能性共有10种(纯合基因型4种:AA、CC、GG、TT;杂合基因型6种:AC、AG、AT、CG、CT、GT)。从每一种真实基因型观察到覆盖该位点的所有碱基的概率,为观察到每一个单独碱基的概率之积,而后者是可以从步骤(2)中建立的概率矩阵中查到的。这样,我们就得到了每一种潜在可能的基因型得到此位点的观察碱基的似然概率(likelihood)。考虑参考基因组的碱基类型和已知多态性位点的信息,我们可以为每一个潜在的基因型赋予一个先验概率,与likelihood结合,得到后验概率。后验概率最高的基因型则为此基因组位点最有可能正确的基因型,即一致序列基因型。该基因型正确的概率,为其后验概率在所有10个基因型后验概率之和中所占的比例。我们将对基因型估计的正确率转换为一个质量分数。
(4) 鉴定单核甘酸多态性位点
将步骤(3)所构建的待测基因组的一致序列中与参考基因组序列不一致的位点从结果中抽出,作为潜在的多态性位点。通过对质量分数设定阈值,将错误率降低至1%以下。
四、具体实施形式
1. 计算最优基因型
我们用贝叶斯公式依据给定的等位基因类型和在染色体上的对应位置的质量值来推断基因型,图1描述了这个方法的具体实现步骤。本方法所需要的输入数据是由Illumina测序技术给出的测序片段(reads)。该方法使用的所有的reads都是都是在参考基因组序列唯一比对上的(例如,将一个亚洲个体基因组序列数据以NCBI人类基因组作为参考序列所做的比对结果),并用这些序列构建待测基因组的一致序列。首先,将序列的质量分数,也就是每个碱基测序错误率的估计值,通过唯一比对上reads进行统计,然后校正,使之更加准确。然后计算基因组每个位置上各种基因型的似然值,然后通过贝叶斯公式计算每种基因型的后验概率,从而确定最有可能的基因型,最后用秩和检验进一步消除错误的杂合位点。
(1) 基因型的先验概率估计
给定一个可用的参考基因组,可以估计待分析的基因组序列相对于参考基因组的突变率。例如,人类基因组上绝大部分位置每代发生突变的概率在
10-8数量级,而两个个体和其共同祖先的差异大约要经过10000代的时间形成。据此推算出来的两个人的单倍染色体SNP概率大约为0.001。每个单倍体跟参考序列大概有1000个核甘酸的差异。假设人的参考基因组序列有0.00001的错误率,则此错误率相对于真实多态性位点的发生率可以忽略。综合这些数据,对于双倍体我们设发生纯合SNP的概率为0.0005,杂合子为0.001。
根据以往对NCBI dbSNPs的研究,转换突变发生的频率是颠换的4倍,但是在这两种变异中各类型之间却几乎是等频率出现的。由此,在我们的SNP 检测模型中用到了这些比率。
例如,假如参考序列上某位点基因型为G,单倍体碱基类型可能是A,C,和T的先验概率都是6.67E-4,G的概率是0.999,而对于双倍体出现GG 组合的概率是0.9985,AA是3.33E-4,TT是8.33E-5,AC和AT是1.11E-7,GC和GT是1.67E-4,AG是6.67E-4,CT是2.78E-8(表1)。
表 1. 二倍体基因组的先验概率。假定参考序列基因组碱基型为脱氧鸟甘酸(G)
A C G T
A 3.33E-4 1.11E-7 6.67E-4 1.11E-7
C 8.33E-5 1.67E-4 2.78E-8
G 0.9985 1.67E-4
T 8.33E-5 (2) 用质量分数进行似然估计
每个位置上的候选等位基因型D可以从与参考序列完全比对上的reads观察出来。假设每种碱基类型T i的可能性为P(D|T i),影响P(D|T i)主要因素有四个,包括 a. 碱基类型,b. 测序质量,c. read 上的位置,d. 出现的次数。所有这些因素都被我们考虑在了本方法的似然估计模型中。
测序过程中的错误和短片段的比对错误都可能引起待测序列与参考序列之间差异的出现。既然下一代的测序片断长度很短,那么会有更多的reads 出现比对错误,因此就出现了错误的SNP。通过设置reads支持最低数目阈值能够滤掉绝大部分的错配的碱基型。测序中,reads靠近3’端低质量的区域,错误通常都不是随机的:AC和GT错配在统计上显著地(p<0.0001)高于其它错配。为解决这个问题,我们把reads坐标,质量分数以及错误的偏向性均考虑在内,用多维数组校正质量分数。然后用这些经过校正的质量分数进行基因型的似然估计。为了避免错误之间的相关性,本方法对PCR扩增所得的大量拷贝进行了质量罚分,以减少其对基因型估计的影响。
2. 测序片段比对的准确性
为了评价read覆盖的准确度和唯一性,我们以人类参考基因组12号染色体作为参考序列,模拟了不同长度的测序片段(包含了0.001比例的SNP和测序错误),然后将这些reads重新比对到人类全基因组参考序列中去。当reads定位到参考序列上具有最少碱基错配的位置时,我们认为是最佳匹配。如果一个read在参考序列上只有一个最佳匹配位置,则称此匹配称为唯一匹配。有多个位置是最佳匹配(同一个read在参考序列上能够和多个位置以同样数目的错配数的比对上)时,我们认为它是重复比对。
从这些数据中我们可以计算出具有唯一比对reads的百分比。对于单向测序唯一比对上的reads,其唯一比对的比例在测序长度从15bp增长到25bp 时变化较大,之后再增加测序长度时,唯一比对的比例仅有很少的变化。78.6%的25bp长度的reads 和91.5%的50bp的reads能够唯一地比对到基因组上。Illumina测序技术典型的测序长度是35bp,在比对结果中发现,没有错误的reads有85.7%能够唯一地比对到基因组上,1个错配的reads有86.3%,两个错配的reads有85.9%。模拟数据的比对结果与炎黄一号基因组实测比对结果相似。从分析中,我们发现,用双向测序技术能够很大的提高唯一比对的reads比例。双向测序技术插入片段长度在100bp 至10kbp(±10%)范围内,能够唯一比对的reads比例随着插入片段长度的增加而增加;其中,在插入片段为200bp的时候有95.4%的成对reads能够唯一的比对上。
用短序列测序片段去比对的话,reads里面包含的SNP和一些测序错误都有可能导致reads比对到不正确的位置上。对于模拟的reads,由于原始的位置事先知道,所以我们能够估计不能比对正确的reads的含量。长度为25bp的单向测序reads,2.3%的具有1个测序错误的reads和3.5%的具有2个测序错误的reads没能正确的匹配上(图2C)。如果测序长度为50bp,这两个比例分别降到了0.6%和0.8%。同理模拟了含有一个测序错误的双向测序reads,插入片段从100bp-10kbp,分别有0.4%和0.06%的reads比对错误;具有2个测序错误的reads 在同样的插入片段下有0.3%和0.06%的不能比上。参见(图2D)
我们从模拟数据中计算了碱基的错误率以检测错误识别的SNPs。至少95%的错误碱基在单向测序reads(35bp)和双向测序reads(插入片段200bp)中都只出现了一次。基于这些数据,我们设置了一个阈值滤除所有低频率的错误碱基(通常设为4)。用这个方法在炎黄一号基因组上查找SNPs,结果只有大约0.036%的错误等位基因没有被检出。在随机DNA 片段的模拟中,36倍测序数据大下约有0.008%的
测序错误未被滤除。为了区分具有高频碱基型的错误和真实的杂合多态性位点,我们利用二项分布(P=0.0001)来检测这两种碱基型的差别,发现剩余的错误碱基型中87.3%都被滤除了。总共,99.93%的由于reads测序错误导致的错误碱基型都被滤除了。
除此之外还有一种导致SNP错误的因素是reads中含有插入删除,这种错误跟reads的比对方式有关。由于SNPs数量大概是小片段的插入删除的5~10倍,比对中优先考虑了不加gap的比对情况,这类含有插入删除的reads可能比对失败。我们允许最长不超过3bp的插入或删除,通过这种方式比对出确实含有插入删除的reads。我们模拟了10000个小片段插入删除来评价SNPs检测的潜在影响,结果发现0.6%的包含插入删除的reads没有检测出来。如果我们要求至少4个reads支持才可信的话,只有3(0.03%)个错误SNP等位基因被检出。
3. Illumina测序质量分数的校正
测序错误的累积导致测序片段3’端错误率要远高于5’端。Illumina测序也能给出一个质量打分,但是这个质量分数是通过信号强度来计算的,并不准确代表错误率的发生。为了纠正这个问题,我们对炎黄一号基因组测序的结果进行了评估,设计了一种方法根据比对错配率来校准质量分数。在这些reads中,真正的SNPs(发生率0.001)和测序错误(发生率0.01)混在一起。在校正中为了尽可能的避免SNPs造成的干扰,我们把已知的SNP位点排除在外。
Illumina测序技术通过每个测序循环的数据来校准质量分数。经过校准后的质量分数跟实际的错配率有一定的差异,而且这个差异是随着read坐标的不同上下波动的(图3A)。目前我们对质量分数的校正仍然是通过比对信息和原始序列信息或者经过Illumina校正后的分数得来。
除了测序误差随着测序循环的不断增加对质量分数造成影响之外,测序仪器本身对碱基的检测也影响了质量分数。Illumina测序技术利用两种不同频率的激光照射四种被标记过的碱基,A,C用一种激光表示,G,T用另一种激光表示。所以AC, GT 测序错误出现的频率就高于其它的错配。我们发现AC, GT错配的概率要比我们模拟比对的情况分别高出58%和72%,同时CG错配概率大约要降低36%(图3B)。例如,质量分数为10(理论错误率0.1)的碱基中,reads与参考序列之间能够观察到的错配AC, CA, GT, TG分别为4.62%,5.27%,5.29%,4.62%,而其它的错配类型仅有1.62%~2.48%。鉴于此结果,我们也按错配类型校正了质量分数。
4. 重复序列的罚分策略
由于DNA建立文库过程中使用了PCR扩增,给定很少数量的DNA起始片断,能够产生大量的拷贝,因此我们能够获取大量一致长度的DNA片断。然而,这些大量复制的片断对测序过程中的随机性有很大影响,有些区域的测序深度不是很理想。这也可能导致各种易与杂合等位基因位点混淆的错误的出现。特别是DNA的损伤被PCR扩增以后可能带来的冗余序列覆盖度,这些可重复的错误很难和多态性位点区分开。因此我们设置了针对扩增重复的罚分规则。如果DNA文库和测序过程都是随机的,那么序列的起点的分布也应该是服从泊松分布。炎黄一号基因组测序的深度是36倍,使用reads长度是35bp,大约0.39%的染色体上的位置有超过6个测序片段起始;然而,理论上这个比例应该是仅有0.07%。因此我们根据一个经验公式来减少有着共同起始位置的reads的影响。我们使用Illumina 1M BeadChip对炎黄一号样品做了基因分型,并检验了纯合的位点。理论上,经过调整后测序错误出现的频率符合泊松分布。(图4)
5. 人类基因组深度测序的结果评估
我们用炎黄一号个人基因组36X测序数据测试了以上方法,在Illumina 1M BeadChip上使用同一DNA样本对一致序列进行基因分型。假设所有的基因分型都是正确的,我们可以通过过识别和欠识别来分类基因分型和测序不一样的位点。所谓欠识别就是在等位基因查找过程中少找了一个杂合位点,过识别就是指多找了一个不正确的碱基类型。
图5显示的是一致序列在参考序列上的覆盖度,Illumina 1M BeadChip分型位点的覆盖度和一致序列的错误率。如果没有质量过滤,整个基因组的覆盖度要低于基因分型位点的覆盖度。因为基因分型位点与基因组的特征是有差别的。增加Q0~Q40的质量限制,基因分型位点的覆盖度有微小的减少:从98.98%降到98.75%;但是当临界值设的更高时,基因分型位点的覆盖度减少得就比较明显。这个可以用SNP位点的先验概率来解释,结果是相对于其它位点质量分数低了很多。过识别和欠识别的概率随着质量阈值的增加
欠识别:0.046%Q0~0.024%Q20,
过识别:0.096%Q0~0.067%Q20
而连续的减少。根据这些数据得到一个质量阈值为Q20,以平衡覆盖度和欠识别过识别几率。
我们也使用了五个其它的过滤步骤用来去除一些共有序列中不可靠的部分:1)要求单倍体最少两个reads,双倍体场染色体最少4个reads覆盖;2)整体深度,包括重复比对上的reads的覆盖,必须小于100;3)局部序列在基因组上的重复数少于2;
4)至少一个双向测序片段支持;5)SNPs间隔至少5bp以上。
(1) 单倍体染色体X
我们对只含有一个X 染色体的雄性基因组进行了测序,因此X 染色体的一致序列查找与单倍体基因组是相同的。每个位点有四种不同的可能的基因型。在X 染色体的37933个Illumina 1M BeadChip 基因分型位点中,99.59%的位点能够被很好的覆盖,而且基因分型和测序有99.96%的共同性(表2)。一致序列在参考基因组中的X 染色体上有88.07%的覆盖度。未有效覆盖的染色体区域是高度重复的,并且几乎没有能够唯一比对的read 。Y 染色体主要由重复序列组成并且参考序列组装得不是很好,因此我们不对其进行讨论。 (2) 双倍体常染色体
为了评价一致序列和SNP 检测的准确性,我们将炎黄一号所有常染色体组装好的一致序列和NCBI 参考序列作了比较,发现一致序列在参考序列的常染
色体有92.25%的覆盖,
在Illumina 1M BeadChip 基因分型位点上有99.22%的覆盖,其中大约有99.92%位点是一致的(表2)。纯合基因分型位点,有0.062%被测序认为是杂合体。
表 2. 本方法所构建的一致序列在基因分型位点的覆盖度
和准确性。
为评估本方法对SNP 的识别准确性,针对部分
过识别的SNPs 采用PCR 扩增后再运用传统的Sanger 测序技术进行测定。在57个测试的中,49(86.0%)个碱基型和芯片得出的结果是一致的。 综上所述,我们的SNP 检测方法和基因分型芯片的结果有很高的一致性,并且 对于过识别的位点,GA 测序技术精确度更高。
五、 附图说明
图1. 大规模平行测序仪一致序列构建的方法框架
图2. 测序片段比对的准确性和唯一性
基因分型的碱基型 分型 位点数 覆盖度 相同 过识别 欠识别 纯合非突变 27,196 99.75% 99.99% 0.007% - 纯合突变 10,737 99.14% 99.87% 0.132% - X 染色体
总数 37,933 99.58% 99.96% 0.042% - 纯合非突变 540,878 99.69% 99.94% 0.062% - 纯合突变 208,436 99.26% 99.78% 0.222% - 杂合
250,667
98.18% 99.99% 0.013% 0.103% 常染色体
总数l 999,981
99.22%
99.92%
0.083%
0.025%
图3. 测序质量分数的不准确和错配的偏向性
图4. 校正前后的起点数目分布
图5. 在不同质量阈值下全基因组覆盖度,基因分型位点的
覆盖度和错误率
图 6. 在不同测序深度下(a)基因分型位点覆盖度(b)欠识别
和过识别的几率
几种常见的基因测序技术的优缺点及应用
随着人类基因组计划的完成,人类对自身遗传信息的了解和掌握有了前所未有的进步。与此同时,分子水平的基因检测技术平台不断发展和完善,使得基因检测技术得到了迅猛发展,基因检测效率不断提高。从最初第一代以 Sanger 测序为代表的直接检测技术和以连锁分析为代表的间接测序技术,到 2005 年,以 Illumina 公司的 Solexa技术和 ABI 公司的 SOLiD 技术为标志的新一代测序(next-generation sequencing,NGS) 的相继出现,测序效率明显提升,时间明显缩短,费用明显降低,基因检测手段有了革命性的变化。其技术正向着大规模、工业化的方向发展,极大地提高了基因检测的检出率,并扩展了疾病在基因水平的研究范围。2009 年 3 月,约翰霍普金斯大学的研究人员在《Science》杂志上发表了通过 NGS外显子测序技术,发现了一个新的遗传性胰腺癌的致病基因PALB2,标志着 NGS 测序技术成功应用于致病基因的鉴定研究。同年,《Nature》发表了采用 NGS 技术发现罕见弗里曼谢尔登综合征MYH3 致病基因突变和《Nat Genet》发表了遗传疾病米勒综合征致病基因。此后,通过 NGS 技术,与遗传相关的致病基因不断被发现,NGS 技术已成为里程碑式的进步。2010 年,《Science》杂志将这一技术评选为当年“十大科学进展”。 近两年,基因检测成为临床诊断和科学研究的热点,得到了突飞猛进和日新月异的发展,越来越多的临床和科研成果不断涌现出来。同时,基因检测已经从单一的遗传疾病专业范畴扩展到复杂疾病和个体化应用更加广阔的领域,其临床检测范围包括高危疾病的新生儿筛查、遗传疾病的诊断和基因携带的检测以及基因药物检测用于指导个体化用药剂量、选择和药物反应等诸多方面的研究。目前,基因检测在临床诊断和医学研究的应用正越来越受到医生的普遍重视和引起研究人员的极大的兴趣。 本文介绍了几种 DNA 水平基因检测常见的方法,比较其优缺点和在临床诊断和科学研究中的应用,对指导研究生和临床医生课外学习,推进临床科研工作和提升科研教学水平有着指导意义。 1、第一代测序 1.1 Sanger 测序采用的是直接测序法。1977年,Frederick Sanger 等发明了双脱氧链末端终止法,这一技术随后成为最为常用的基因测序技术。2001 年,Allan Maxam 和 Walter Gibert 发明了 Sanger 测序法,并在此后的 10 年里成为基因检测的金标准。其基本原理即双脱氧核苷三磷酸(dideoxyribonucleoside triphosphate,ddNTP) 缺乏PCR 延伸所需的 3'-OH,因此每当 DNA 链加入分子 ddNTP,延伸便终止。每一次 DNA 测序是由 4个独立的反应组成,将模板、引物和 4 种含有不
ACMG全外显子测序指南 摘要:美国医学遗传学与基因组学学会(ACMG)以前为序列突变的解释提供了指导.1在过去十年中,随着高通量测序的出现,测序技术迅速发展。通过采用和利用下一代测序,临床实验室正在进行基因分型,单基因,基因组,外显子,基因组,转录组和遗传疾病表观遗传学检测的不断增加的遗传检测目录。由于复杂性增加,基因检测的这种转变伴随着序列解释的新挑战。在这方面,ACMG于2013年召集了一个由ACMG,分子病理学协会(AMP)和美国病理学家学会的代表组成的工作组,重新审视和修订了序列突变解释的标准和准则。该组由临床实验室主任和临床医生组成。本报告代表ACMG,AMP和美国病理学家利益相关者联盟组成的工作组的专家意见。这些建议主要适用于临床实验室使用的遗传检测的范围,包括基因分型,单基因,panel,外显子和基因组。本报告建议使用具体的标准术语- “致病性”,“可能致病性”,“不确定性意义”,“可能良性”和“良性”来描述在导致孟德尔病症的基因中鉴定的突变。此外,该建议描述了基于使用典型类型的突变证据(例如,群体数据,计算数据,功能数据,分离数据)的标准将突变分类为这五个类别的过程。由于本报告中描述的临床基因检测的分析和解释的复杂性增加,ACMG强烈建议临床分子遗传学检测应在经过临床实验室改进修订批准的实验室进行,结果由相关职业认证的临床分子遗传学家或分子遗传病理学家或同等学科专家进行解释。 关键词:ACMG实验室指导; 临床遗传检测; 解释;报告; 序列变异术语;突变报告 前言 临床分子实验室正在不断增加检测的新的序列突变,因为在检测患者标本时不断发现大量与基因疾病相关的基因。虽然一些表型与单个基因相关,但许多与多个基因相关。我们对任何给定序列突变的临床意义的理解是循序渐进的,其范围从那些几乎肯定是疾病致病性突变到几乎肯定是良性的突变。虽然以前的美国医学遗传学和基因组学会(ACMG)的建议提供了序列突变的解释类别和解释算法,但是这些建议没有提供定义的术语或详细的突变分类指南.1。本报告描述了
基因测序技术的优缺点及应用 随着人类基因组计划的完成,人类对自身遗传信息的了解和掌握有了前所未有的进步。与此同时,分子水平的基因检测技术平台不断发展和完善,使得基因检测技术得到了迅猛发展,基因检测效率不断提高。从最初第一代以 Sanger 测序为代表的直接检测技术和以连锁分析为代表的间接测序技术,到 2005 年,以Illumina 公司的 Solexa技术和 ABI 公司的 SOLiD 技术为标志的新一代测序 (next-generation sequencing,NGS) 的相继出现,测序效率明显提升,时间明显缩短,费用明显降低,基因检测手段有了革命性的变化。其技术正向着大规模、工业化的方向发展,极大地提高了基因检测的检出率,并扩展了疾病在基因水平的研究范围。2009 年 3 月,约翰霍普金斯大学的研究人员在《Science》杂志上发表了通过 NGS外显子测序技术,发现了一个新的遗传性胰腺癌的致病基因PALB2,标志着 NGS 测序技术成功应用于致病基因的鉴定研究。同年,《Nature》发表了采用 NGS 技术发现罕见弗里曼谢尔登综合征MYH3 致病基因突变和《Nat Genet》发表了遗传疾病米勒综合征致病基因。此后,通过 NGS 技术,与遗传相关的致病基因不断被发现,NGS 技术已成为里程碑式的进步。2010 年,《Science》杂志将这一技术评选为当年“十大科学进展”。 近两年,基因检测成为临床诊断和科学研究的热点,得到了突飞猛进和日新月异的发展,越来越多的临床和科研成果不断涌现出来。同时,基因检测已经从单一的遗传疾病专业范畴扩展到复杂疾病和个体化应用更加广阔的领域,其临床检测范围包括高危疾病的新生儿筛查、遗传疾病的诊断和基因携带的检测以及基因药物检测用于指导个体化用药剂量、选择和药物反应等诸多方面的研究。目前,基因检测在临床诊断和医学研究的应用正越来越受到医生的普遍重视和引起研究人员的极大的兴趣。 本文介绍了几种 DNA 水平基因检测常见的方法,比较其优缺点和在临床诊断和科学研究中的应用,对指导研究生和临床医生课外学习,推进临床科研工作和提升科研教学水平有着指导意义。 1、第一代测序 1.1 Sanger 测序采用的是直接测序法。1977年,Frederick Sanger 等发明了双脱氧链末端终止法,这一技术随后成为最为常用的基因测序技术。2001 年,Allan Maxam 和 Walter Gibert 发明了 Sanger 测序法,并在此后的 10 年里成为基因检测的金标准。其基本原理即双脱氧核苷三磷酸(dideoxyribonucleoside triphosphate,ddNTP) 缺乏PCR 延伸所需的 3'-OH,因此每当 DNA 链加入分子 ddNTP,延伸便终止。每一次 DNA 测序是由 4个独立的反应组成,将模板、引物和 4 种含有不同的放射性同位素标记的核苷酸的ddNTP 分别与DNA 聚合酶混合形成长短不一的片段,大量起始点相同、终止点不同的 DNA 片段存在于反应体系中,具有单个碱基差别的 DNA 序列可以被聚丙烯酰胺变性凝胶电泳分离出来,得到放射性同位素自显影条带。依据电泳条带读取DNA 双链的碱基序列。 人类基因组的测序正是基于该技术完成的。Sanger 测序这种直接测序方法具有高度的准确性和简单、快捷等特点。目前,依然对于一些临床上小样本遗传疾病基因的鉴定具有很高的实用价值。例如,临床上采用 Sanger 直接测序 FGFR 2 基因证实单基因 Apert 综合征和直接测序 TCOF1 基因可以检出多达 90% 的
50个全外显子测序揭示人类的高原适应机制 Xin Yi等。 Science 329, 75 (2010); DOI: 10.1126/science.1190371 50个全外显子测序揭示人类的高原适应机制 生活于青藏高原的藏族人表现出了对极端高原环境的遗传适应性。我们对50个藏族人进行全外显子基因组测序,它们包含了92%的人类基因编码序列,人均覆盖度为18倍。基因分析显示了该特殊人群等位基因频率的变化,表明这些人对高原环境具有很强的适应性。 研究显示,表现出最强自然选择信号的基因是编码内皮细胞含PAS结构域蛋白-1(EPAS1)的基因,这是一个参与应答缺氧的转录因子。研究显示,EPAS1基因的一个单核苷酸多态性(SNP)在78%的藏族和汉族人群中存在差异,这是目前发现的速率改变最快的等位基因。该单核苷酸多态性与红细胞丰度的关联分析也支持EPAS1改变在适应缺氧环境中的作用,进一步表明它是适应高原环境的一个重要的遗传位点。 在广袤的大自然中生存的人类可能会存在文化和基因上的适应。其中人类面临的最严厉的环境挑战就是高海拔地区(如青藏高原)的低含氧量。这一地区的许多居民在海拔4000米以上居住,那里的氧气浓度比海平面大约低40%。藏族对缺氧环境有着他们自己的遗传适应性,如出生体重(1),血红蛋白水平(2),婴儿(3)和运动后的成年人(4)血液中的氧饱和度。这些结果暗示了高原适应机制的自然选择历史,我们对整个基因组的遗传差异进行分析,可能会发现这一点。 我们对中国西藏自治区海拔4300米以上(5)的两个村庄里的50个非亲个体进行全外显子基因组测序。针对将近两万个基因的外显子和侧翼区的34Mb序列,利用罗氏NimbleGen公司(威斯康星州麦迪逊市)的2.1M外显子序列捕获芯片(6)将其富集。测序采用了Illumina公司(加利福尼亚州圣地亚哥市)的基因组分析仪II平台,并使用序列比对程序SOAP(7)将测序片段比对到人类参考基因组序列上[美国生物技术信息中心(NCBI) 36. 3版]。 1深圳华大基因研究院,中国深圳,518083。 2中国科学院研究生院,中国北京,100062。 3加州大学伯克利分校综合生物学与统计系,美国加州,94820。4华南理工大学生物系本科创新班,中国广州,510641。 5西藏自治区人民医院,中国拉萨,850000。 6加州大学戴维斯分校进化与生态学系,美国加州,95616。 7哥本哈根大学生物系,丹麦哥本哈根,1165。 8华南理工大学理学院本科创新班,中国广州,510641。 9深圳大学医学院基因组研究所,中国深圳,518060。 10拉萨市人民医院,中国拉萨,850000。 11西藏军区总医院,中国拉萨,850007。 12西双版纳傣族自治州人民医院,中国云南景洪,666100。 *以上机构及相关人员对本研究作出了贡献。如有疑问请联系: E-mail:wangjian@https://www.doczj.com/doc/25463102.html, ( Ji.W.); wangj@https://www.doczj.com/doc/25463102.html, ( Ju.W.); rasmus_nielsen@https://www.doczj.com/doc/25463102.html, (R.N.) 全外显子测序的平均深度为18倍(表S1),但这并不能保证个别基因型的准确性。因此,我们用贝叶斯统计法(5)估算出每个可能的基因型概率,从而估算出单核苷酸多态性(SNP)的概率和每个位点的人类等位基因频率。在藏族样本中总共151825个SNPs有超过50%被识别出是可变的,有101668个超过99%的SNP是可变的(表S2)。Sanger测序验证了56个SNPs 中的53个,至少包含95%的SNP和3%~50%的次等位基因频率。等位基因频率的估算值显示存在过量的低频变异(图S1),特别是在非同义SNPs中。 该数据与来自于北京的40个汉族人的基因组进行比较[样本来自于中测检测(CHB)人类基因组单体型图计划(HapMap),属于1000个基因组计划的一部分(https://www.doczj.com/doc/25463102.html,)],测序得出汉族人均大约4倍的覆盖度。北京的海拔不超过50米,几乎所有的汉族人来自于海拔2000米以下。在较低的遗传分化基础上汉族人样本与藏族人样本形成鲜明的对比(F ST= 0.026)。这两个藏族村庄是体现该族遗传结构的最小单位(F ST= 0.014),因此,我们可以将他们当着整个群体进行分析。我们观察到,汉藏之间的等位基因频率有着很强的协方差(图1),但是过量的SNPs在汉族中频率很低,在藏族中频率中等。 从两个群体同义位点的二维频谱,可以估算出人类历史模型(8)。最佳拟合模型表明,藏族和汉族人群在2750年前出现分化,汉族人口从最初的小规模逐渐增大,藏族人口从最初的大规模逐渐减少(图S2)。这估计是由于藏族人移民至汉族区,双方长期相互渗透所造成的。
附件:全外显子组检测技术参数要求 一、公司资质: 1.拥有先进的高通量二代测序平台和高性能计算平台; 2.具有短期处理大量样本,进行全外显子组和全基因组测序的经验; 3.实验室具有国内或国外权威机构的资质认证; 4.*应标的公司必须通过医学遗传中心选送的样本测试(三个以上生物学重复),并且需交 付原始下机数据,以中心提供的标准化流程统一进行质量评估。 二、技术参数: 1)污染防控 具有独立的实验方法进行样本身份鉴定,可追溯样本间发生的错误 2)测序质量 1.Q20平均比例在90%以上。 2.Q30平均比例在85%以上。 3.GC content 分布无明显偏移。 3)测序深度、覆盖度统计 下文涉及的数据均为经过去接头、比对、排序和去重后的有效数据。数据统计涉及的相关软件除特别说明外,应使用默认参数。 1.数据质量要求: 1)Mapped unique reads相对总reads的比例(PCT_PF_UQ_READS_ALIGNED)不得低于
99% 2)有效数据总量(PF_UQ_BASES_ALIGNED)不得低于10G 3)On targeted bases相对总bases的比例(PCT_USABLE_BASES_ON_BAIT)不得低于50% 4)On and near targeted bases相对总bases的比例(PCT_SELECTED_BASES)不得低于 80% 5)全外显子碱基10X覆盖率(PCT_TARGET_BASES_10X)不得低于95% 6)全外显子碱基30X覆盖率(PCT_TARGET_BASES_30X)不得低于80% 7)全外显子组各区域覆盖的一致性统计要求:80%以上的target region的normalized coverage值不得低于0.3 三、项目内容: 500例耳聋患者全外显子组测序技术服务
人全外显子组序列捕获及第二代测序 概述 外显子组是指全部外显子区域的集合,该区域包含合成蛋白质所需要的重要信息,涵盖了与个体表型相关的大部分功能性变异。外显子组序列捕获及第二代测序是一种新型的基因组分析技术:外显子序列捕获芯片(或溶液)可在同一张芯片上以高特异性和高覆盖率捕获研究者感兴趣的目标外显子区域,后续利用Solexa/SOLiD/Roche 454测序直接解析数据。 与全基因组重测序相比,外显子组测序只需针对外显子区域的DNA 即可,覆盖度更深、数据准确性更高,更加简便、经济、高效。可用于寻找复杂疾病(如:癌症、糖尿病、肥胖症等)的致病基因和易感基因等的研究。同时,基于大量的公共数据库提供的外显子数据,我们能够结合现有资源更好地解释我们的研究结果。 目前,SBC提供的外显子组序列捕获芯片是NimbleGen Sequence Capture 2.1M Human Exome Array及Agilent SureSelect Target Enrichment System(Human Exome)。 技术路线 以Nimblegen外显子捕获结合Solexa测序为例加以说明:基因组DNA首先被随机打断成500bp左右的片段,随后在DNA片段两端分别连接上接头。经过PCR库检合格后的DNA 片段与NimbleGen 2.1M Human Exome Array芯片进行杂交。去除未与芯片结合的背景DNA 后,将经过富集的外显子区域的DNA片段洗脱下来。这些DNA片段又随机连接成长DNA片段
后,再次被随机打断并在其两端加上测序接头,经过LM-PCR的线性扩增,在经qPCR质量检测合格后即可上机测序。 外显子组测序的实验流程示意图(https://www.doczj.com/doc/25463102.html,) 生物信息学分析流程图 研究内容 1.外显子组捕获与测序 将基因组DNA随机打断成片段,通过与人全外显子捕获芯片杂交富集外显子区域,通过第二代测序技术对捕获的序列进行测序。 2.基本数据分析 数据产出统计:对测序结果进行图像识别(Base calling),去除污染及接头序列;统计结果包括:测定的序列(Reads)长度、Reads数量、数据产量。 3. 高级数据分析 高级数据分析内容包括: (1)Clean reads序列与参考基因组序列比对; (2)目标外显子区域测序深度分析; (3)目标外显子区域一致序列组装;
几种常见的基因测序技术的优缺点及应用 发布时间:2014-07-19 来源:毕业论文网 随着人类基因组计划的完成,人类对自身遗传信息的了解和掌握有了前所未有的进步。与此同时,分子水平的基因检测技术平台不断发展和完善,使得基因检测技术得到了迅猛发展,基因检测效率不断提高。从最初第一代以Sanger 测序为代表的直接检测技术和以连锁分析为代表的间接测序技术,到2005 年,以Illumina 公司的Solexa技术和ABI 公司的SOLiD 技术为标志的新一代测序(next-generation sequencing,NGS) 的相继出现,测序效率明显提升,时间明显缩短,费用明显降低,基因检测手段有了革命性的变化。其技术正向着大规模、工业化的方向发展,极大地提高了基因检测的检出率,并扩展了疾病在基因水平的研究范围。2009 年 3 月,约翰霍普金斯大学的研究人员在《Science》杂志上发表了通过NGS外显子测序技术,发现了一个新的遗传性胰腺癌的致病基因PALB2,标志着NGS 测序技术成功应用于致病基因的鉴定研究。同年,《Nature》发表了采用NGS 技术发现罕见弗里曼谢尔登综合征MYH3 致病基因突变和《Nat Genet》发表了遗传疾病米勒综合征致病基因。此后,通过NGS 技术,与遗传相关的致病基因不断被发现,NGS 技术已成为里程碑式的进步。2010 年,《Science》杂志将这一技术评选为当年“十大科学进展”。 近两年,基因检测成为临床诊断和科学研究的热点,得到了突飞猛进和日新月异的发展,越来越多的临床和科研成果不断涌现出来。同时,基因检测已经从单一的遗传疾病专业范畴扩展到复杂疾病和个体化应用更加广阔的领域,其临床检测范围包括高危疾病的新生儿筛查、遗传疾病的诊断和基因携带的检测以及基因药物检测用于指导个体化用药剂量、选择和药物反应等诸多方面的研究。目前,基因检测在临床诊断和医学研究的应用正越来越受到医生的普遍重视和引起研究人员的极大的兴趣。 本文介绍了几种DNA 水平基因检测常见的方法,比较其优缺点和在临床诊断和科学研究中的应用,对指导研究生和临床医生课外学习,推进临床科研工作和提升科研教学水平有着指导意义。 1、第一代测序 1.1 Sanger 测序采用的是直接测序法。1977年,Frederick Sanger 等发明了双脱氧链末端终止法,这一技术随后成为最为常用的基因测序技术。2001 年,Allan Maxam 和Walter Gibert 发明了Sanger 测序法,并在此后的10 年里成为基因检测的金标准。其基本原理即双脱氧核苷三磷酸(dideoxyribonucleoside triphosphate,ddNTP) 缺乏PCR 延伸所需的 3'-OH,因此每当DNA 链加入分子ddNTP,延伸便终止。每一次DNA 测序是由4个独立的反应组成,将模板、引物和 4 种含有不同的放射性同位素标记的核苷酸的ddNTP 分别与DNA 聚合酶混合形成长短不一的片段,大量起始点相同、终止点不同的DNA 片段存在于反应体系中,具有单个碱基差别的DNA 序列可以被聚丙烯酰胺变性凝胶电泳分离出来,得到放射性同位素自显影条带。依据电泳条带读取DNA 双链的碱基序列。 人类基因组的测序正是基于该技术完成的。Sanger 测序这种直接测序方法具有高度的准确性和简单、快捷等特点。目前,依然对于一些临床上小样本遗传疾病基因的鉴定具有很高的实用价值。例如,临床上采用Sanger 直接测序FGFR 2 基因证实单基因Apert 综合征和直接测序TCOF1 基因可以检出多达90% 的与Treacher Collins 综合征相关的突变。值得注意的是,Sanger 测序是针对已知致病基因的突变位点设计引物,进行PCR 直接扩增测序。
华大智造外显子捕获测序解决方案 概述 随着测序技术发展和成本降低,临床外显子组测序(cWES)和全外显子测序(WES)在遗传病检测领域崭露头角。外显子测序借助捕获探针(DNA或RNA)对人基因组约1-2%的区域测序,可覆盖绝大多数基因的编码序列和>99%(临床基因组资源库,ClinGen)疾病相关区域。华大智造基于自有的探针合成平台和高通量测序仪(MGISEQ/BGISEQ 系列),能为客户提供外显子测序一站式解决方案。 图1 外显子测序示意图(以MGI测序平台为例)
MGIEasy 外显子组捕获V5探针试剂套装 MGIEasy 外显子组捕获V5探针试剂套装除了涵盖传统外显子探针覆盖的区域,还有针对性的做了探针优化,保证了生育健康、新生儿、心脑血管、遗传性肿瘤、单基因病、安全用药、个人基因组、遗传性耳聋、免疫缺陷、线粒体缺陷等致病基因的全覆盖。 产品亮点 ●探针区域69Mb ●更多的疾病致病位点 ●更优的数据利用率 ●稳定而高效的捕获效率 技术优势 数据库覆盖情况 MGI V5与竞品(Vendor A6/N3/I)比,有更多的独有区域,涵盖了华大自主研发的 图2 CCDS、GENCODE、UCSC、miRBase和RefSeq数据库基因数量覆盖情况 基因覆盖更全面
MGI V5能100%覆盖的基因数达到455个,远高于A5 (125个)、N3 (33个)和I (357个),其独有100%覆盖基因数达到160个,是A5和N3之和。 BBS10基因是巴比二氏综合征的致病基因,MGI V5完整涵盖了基因区和内含子区,其中包括ClinVar数据库中报道的已知临床突变位点。 基因覆盖均一性更优 MGI V5在测序深度达到100x时,96%的区域覆盖度均能达到20X以上。与竞品N3和I共有的区域,MGI V5显示了更优秀的覆盖均一性。 性能比较 图3 100%覆盖的基因数和BBS10基因覆盖情况 图4 >96%区域达到20X覆盖图5 共有区域的覆盖更均一
人外显子测序 药明康德基因中心,陆桂1. 什么是外显子测序(whole exon sequencing)? 外显子组测序是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。外显子测序相对于基因组重测序成本较低,对研究基因的SNP、Indel 等具有较大的优势,但无法研究基因组结构变异如染色体断裂重组等。 2. 外显子捕获试剂盒有哪些? 目前主要有Roche、Illumina和Agilent三家的外显子捕获试剂。Nimblegen和Illumina的捕获试剂盒中的探针是DNA探针,化学性质稳;Agilent的捕获试剂盒是RNA探针,有可能RNA 不是很稳定。 3. 外显子捕获效率是什么? 外显子测序过程中要用到杂交过程。在人的染色体上有许多与外显子有同源性的部分,这些有同源性的部分很可能在杂交过程中也被捕获下来。所以,测到的序列中,有一部分不是外显子序列。我们把测序得是外显子的部分占全部测序序列的比列称为捕获效率。 Nimblegen大约是70% Agilent大约是60% Illumina大约是50% 4. 外显子测序一般建议做多少倍的覆盖? 一般做100X或者150X。较高的覆盖倍数,对于测异质性的遗传变质,可以发现小比例的突变。另外,外显子测序的覆盖不是很均匀,这样较高的平均覆盖率有利于保证大部分的区域有足够的覆盖倍数。 5. 外显子测序能够测出多大的片段缺失? 大致能测出50bp的片段缺失。目前的测序主要还是用Hiseq 2000,单侧的测长就是100bp。由于外显子测序的覆盖很不平均,所以如果有大段的缺失,无法判断是因为杂交没有捕获到,还是因为缺失。目前能够测到的,就是在一个read中发现的缺失。一个read的长度也就是100bp,所以大到50bp以下的片段缺失可以从外显子测序中测出来。 6. 外显子捕获可以做CNV吗? 外显子测序因为有一个杂交捕获的过程,这样就会有一个杂交捕获效率的问题。各个外显子的杂交效率是不同的,其同源竞争的情况也不同,所以不同的外显子的覆盖率的差异就很大。所以一般情况下,外显子测序不能用于CNV的检测。但在癌症研究中,利用癌组织和癌旁组织对照,可以检测CNV。 现在我们有另外两种常规方法来检测CNV,一种是全基因组重测序,另外一种是用Affymetrix SNP6.0的芯片来测。其中Affymetrix SNP6.0的检测费用大约只有全基因测序费用的1/10,是一个相对经济的手段。 7. 外显子测序的优点是什么?
外显子捕获结题报告2010-11-22
内容 1 项目信息 (1) 2 工作流程介绍 (2) 2.1 Agilent液相捕获平台 (2) 2.2 NimbleGen 液相捕获平台 (3) 2.3 生物信息分析流程 (4) 3 分析报告 (5) 结果 (5) 3.1 标准生物信息分析 (5) 3.1.1 数据产出统计 (5) 3.1.2 目标区域单碱基深度分布图 (6) 3.1.3外显子捕获测序的均一性 (7) 3.1.4一致序列组装和SNP检测 (7) 3.1.5 SNP注释 (8) 3.1.6插入/缺失(indels)检测 (9) 3.1.7插入/缺失(indels)注释 (9) 3.2个性化分析 (9) 3.2.1氨基酸替换预测 (9) 3.2.2群体SNP检测和等位基因频率估计 (12) 3.2.3孟德尔遗传病分析 (13) 3.2.4 NGS-GW AS 分析 (14) 3.2.5正向选择信号的检测 (14) 4 数据分析方法说明 (15) 4.1信息分析软件及常用参数介绍 (15) 4.2参考数据库 (16) 4.3数据文件格式 (17)
1 项目信息 PROJECT NAME CONTRACT NUMBER SAMPLE INFORMATION Species Information Genome Information Additional Information CUSTOMER INFORMATION PI Contact Person Company Name Contact Methods Name Tel E-mail Name Tel E-mail CONTACT INFORMATION (BGI) Sales Information Name Tel E-mail Name Tel E-mail Customer Service Name Tel E-mail Name Tel E-mail PROJECT DIRECTOR APPROVAL THE RESULTS HAVE BEEN APPROVED AND CAN BE SUBMITTED Signature: Date:
技术参数 样品要求捕获平台测序策略 测序深度 项目周期 外显子组测序 37天 1. 单基因病/复杂疾病有效测序深度50X以上 2. 肿瘤有效测序深度100X以上 注:可根据老师研究目的进行更高深度测序 HiSeq PE150 Agilent SureselectXT Custom Kit 样品总量:≥1.0 μg DNA (提取自新鲜及冻存样本) ≥1.5 μg DNA (提取自FFPE样本)样品浓度:≥20 ng/μl 参考文献 外显子组测序(Whole Exome Sequencing,WES)是利用探针杂交富集外显子区域的DNA序列,通过高通量测序,发现与蛋白质功能变异相关遗传突 变的技术手段。相比于全基因组测序,外显子组测序更加经济、高效。 1. 直接对蛋白编码序列进行测序,找出影响蛋白结构的变异 2. 高深度测序,可发现常见变异及频率低于1%的罕见变异 3. 针对外显子组区域测序,约占基因组的1%,有效降低费用,周期和工作量 技术优势 生物信息分析 基本信息分析 1. 数据质控:去除接头污染和低质量数据 2. 与参考序列进行比对、统计测序深度及覆盖度 3. SNP/InDel检测、注释及统计 4. Somatic SNV/InDel检测、注释及统计(成对样本) 高级信息分析(单基因病) 高级信息分析(复杂疾病) 高级信息分析(癌症) 1. 突变位点过滤 2. 显/隐性遗传模式分析(需老师提供家系信息) 2.1. 显性遗传模式分析 2.2. 隐性遗传模式分析 3. 候选基因功能注释 4. 新生突变筛选及分析(成三/成四家系) 4.1. de novo mutation 筛选 4.2. 新生突变速率计算 5. 候选基因功能富集 6. 蛋白互作网络分析(PPI) 7. 基因显著性分析 (推荐20对Case/Control or trios样本) 1. 突变位点过滤 2. 显/隐性遗传模式分析(需老师提供家系信息) 2.1. 显性遗传模式分析 2.2. 隐性遗传模式分析 3. 候选基因功能注释 4. 基因功能及通路分析 5. 家系连锁分析 6. 纯合子区域(ROH)分析 1. 易感基因筛查 2. NMF突变特征及突变频谱分析 3. 已知驱动基因筛选 4. 高频突变基因统计及通路富集分析 5. MRT高频突变基因相关性分析 6. OncodriveCLUST驱动基因预测 7. 高频CNV分布及重现性分析 8. 肿瘤纯度/倍性分析 9. 异质性/克隆结构分析 10. NovoDrug高频突变基因靶向用药预测11. NovoDR耐药突变筛选12. 基因组变异Circos图展示 案例解析 [案例一] 单基因病研究:外显子测序解析卵巢早衰的遗传因素[12] 卵巢早衰通常是指女性40岁之前闭经,1%的妇女患有此病,病因复杂,被认为受到遗传因素的影响。这项研究利用外显子测序技术首次在中东家系1(MO1DA)的卵巢早衰病人中发现了减数分裂基因中的STAG3基因突变可以导致隐性遗传卵巢早衰,也在小鼠动物模型和卵巢早衰病患中得到了证实。为探索卵巢早衰或卵巢功能不全的发生机理,以及阐明该病的临床高度异质性和遗传病因复杂性开辟了一个新的研究途径。 [案例二] 复杂疾病研究:外显子测序鉴定肌萎缩性脊髓侧索硬化症(ALS)的致病 基因[13] 肌萎缩性脊髓侧索硬化症(ALS),又称为渐冻症,是一种成年型的神经退行性疾病。本研究选取了47个父母+患病儿的ALS家系,利用全外显子测序寻找De novo mutatio n 。发现了25个de novo突变基因,进行功能聚类分析,锁定了1个与染色质包装、神经树突生长相关的基因CREST,后期通过细胞试验验证了该基因突变会影响神经元的伸展,证实CREST突变与ALS相关。 [案例三] 癌症研究:外显子测序研究局限性肺腺癌瘤内异质性[14] 本研究采用多区域取样分析瘤内异质性的研究思路,对11位患者的局限性肺腺癌的48个肿瘤样品进行了外显子测序。共鉴定出7269个体突变,其中21个是已知的与癌症相关的基因突变,76% 的体突变及21个已知癌症基因突变中的20个都可以在同一肿瘤的所有区域样品中检测到,表明对肿瘤的某一区域进行单次活检,以适当的深度对其测序,可以鉴别出绝大多数突变。而前期关于肾透明细胞癌的研究结果表明,肿瘤不同区域样品的共有突变仅占突变总数的31%~37%,说明肿瘤异质性在不同癌种间存在差异。 [1] Krawitz PM, Schweiger MR, R?delsperger C, et al. Identity-by-descent filtering of exome sequence data identifies PIGV mutations in hyperphosphatasia mental retardation syndrome[J]. Nature Genetics, 2010, 42(10): 827-829.[2] Liu Y, Gao M, Lv YM, et al. Confirmation by exome sequencing of the pathogenic role of NCSTN mutations in acne inversa (hidradenitis suppurativa) [J]. Journal of Investigative Dermatology,2011, 131(7): 1570-1572. [3] Wei A H, Zang D J, Zhang Z, et al. Exome sequencing identifies SLC24A5 as a candidate gene for nonsyndromic oculocutaneous albinism[J]. Journal of Investigative Dermatology, 2013, 133(7): 1834-1840. [4] Sanna-Cherchi S, Sampogna R V, Papeta N, et al. Mutations in DSTYK and dominant urinary tract malformations[J]. New England Journal of Medicine, 2013, 369(7): 621-629.[5] Musunuru K, Pirruccello J P , Do R, et al. Exome sequencing, ANGPTL3 mutations, and familial combined hypolipidemia[J]. New England Journal of Medicine, 2010, 363(23): 2220-2227. [6] O'Roak B J, Deriziotis P , Lee C, et al. Exome sequencing in sporadic autism spectrum disorders identifies severe de novo mutations[J]. Nature genetics, 2011, 43(6): 585-589. [7] Jones S, Wang T L, Shih I M, et al. Frequent mutations of chromatin remodeling gene ARID1A in ovarian clear cell carcinoma[J]. Science, 2010, 330(6001): 228-231. [8] Yan X J, Xu J, Gu Z H, et al. Exome sequencing identifies somatic mutations of DNA methyltransferase gene DNMT3A in acute monocytic leukemia[J]. Nature Genetics, 2011, 43(4): 309-315. [9] Rudin C M, Durinck S, Stawiski E W, et al. Comprehensive genomic analysis identifies SOX2 as a frequently amplified gene in small-cell lung cancer[J]. Nature Genetics, 2012, 44(10): 1111-1116. [10] Yi X, Liang Y, Huerta-Sanchez E, et al. Sequencing of 50 human exomes reveals adaptation to high altitude[J]. Science, 2010, 329(5987): 75-78. [11] Tennessen J A, Bigham A W, O’Connor T D, et al. Evolution and functional impact of rare coding variation from deep sequencing of human exomes[J]. Science, 2012, 337(6090): 64-69. [12] Caburet S, Arboleda V A, Llano E, et al. Mutant cohesin in premature ovarian failure[J]. New England Journal of Medicine, 2014, 370(10): 943-949.[13] Chesi A, Staahl B T, Jovicic A, et al. Exome sequencing to identify de novo mutations in sporadic ALS trios[J]. Nature Neuroscience, 2013, 16(7): 851-855.[14] Zhang J, Fujimoto J, Zhang J, et al. Intratumor heterogeneity in localized lung adenocarcinomas delineated by multi region sequencing[J]. Science, 2014, 346: 256-259. 群体研究 藏族人高原适应性研究[10];深度解析人类罕见遗传变异[11];…… 图1 STAG3 基因结构图 (红色箭头为 STAG3 基因突变位置) 图2 ALS家系图及CREST突变功能验证 图3 产生化疗抗性的个体样本中体突变的数量及频率
外显子组测序 介绍 外显子(exon)是真核生物基因的一部分,包含着合成蛋白质所需要的信息。全部外显子被称为“外显子组”(Exome)。外显子组测序(Exome sequencing)是指利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。由于外显子组测序捕获目标区域只占人类基因组长度的约1%,因此远比进行全基因组序列测序来得更简便、经济,目标区域覆盖度也更高,便于变异检测。 该项技术可用于以下研究 1)检测疾病样本中外显子区域内高风险碱基变异位点; 2)配合大样本分析,确定孟德尔遗传疾病相关外显子SNP位点和基因; 3)在癌症研究过程中,检测癌症样本外显子区域内的体细胞突变位点和潜在的融合基因; 4)用于种群遗传学研究的大规模样本基因组分析,检测SNP位点、LD并绘制种群图谱。 我们能提供详尽的全基因组重测序数据的处理和分析服务。 如您没有标准化的数据、只需流程中的局部分析内容或要求特立独行的数据分析思路,我们亦能满足您的要求。 数据处理和分析流程图
预期结果示例图 示例图1 各类型SNV在样本中的个数统计。 示例图2 不同类型外显子区域上的SNV类型统计。 示例图4 融合基因预测[1]
示例图4 大量样本的GWAS分析结果[2] 示例图5 肿瘤样本高频率突变基因统计[3] 示例图来源文献 [1]. Kangaspeska, S., et al., Reanalysis of RNA-sequencing data reveals several additional fusion genes with multiple isoforms. PLoS One, 2012. 7(10): p. e48745. [2]. Craig, J.E., et al., Rapid inexpensive genome-wide association using pooled whole blood. Genome Res, 2009. 19(11): p. 2075-80.
2019年一2月第39卷一第2期 基础医学与临床Basic&ClinicalMedicineFebruary2019Vol.39一No.2收稿日期:2017 ̄11 ̄09一一修回日期:2018 ̄03 ̄27 基金项目:湖北省教育厅重点项目(D20171205)?湖北省自然科学基金(2017CFB455) ?通信作者(correspondingauthor):zrt0116@126.com文章编号:1001 ̄6325(2019)02 ̄0272 ̄05短篇综述一 全外显子组测序在肺癌的发病机制研究和诊治中的临床意义 唐永莉?张瑞涛? (三峡大学医学院?湖北宜昌443000) 摘要:全外显子组测序(WES)是利用序列捕获技术将全外显子区域DNA捕捉并富集后进行高通量测序的基因分析方法?外显子组测序较全基因组序列测序更简便二经济和高效?其目标区域覆盖度也更高?便于变异检测?外显子组测序技术已经应用到寻找与各种复杂疾病相关的致病基因和易感基因的研究中?肺癌是常见的恶性肿瘤之一?基于国内外对全外显子测序在肺癌中的研究成果?现就全外显子测序在肺癌的诊治以及肺癌的发生机制的研究进行综述? 关键词:全外显子组测序?肺癌?易感基因?基因突变 中图分类号:R734 2一一文献标志码:A Clinicalsignificanceofwholeexomesequencinginmechanismresearchandtreamentinlungcancer TANGYong ̄li?ZHANGRui ̄tao? (MedicalCollegeofChinaThreeGorgesUniversity?Yichang443000?China)Abstract:Wholeexomesequencing(WES)isageneanalysismethodthatusesthesequencecapturetech ̄niquetocaptureandenrichthewholeexonregionDNAandtoperformhigh ̄throughputsequencing.WEScanbeusedtodetectgeneticmutationsassociatedwithproteinfunctionalvariabilitydirectly.SincetheWESisse ̄quencedonlyfortheDNAoftheexonregion?itismuchsimple?moreeconomicalandefficientthanthewholegenomesequencing?anditstargetareacoverageishigher?whichiseasytodetect.Atpresent?WEShasbeenappliedtothestudyofpathogenicgenesandsusceptibilitygenesassociatedwithvariouscomplexdiseasessuchaslungcancer.Inthispaper?TheclinicalsignificanceofWESinmechanismresearchandtreamentinlungcancerwasbeenreviewed.Keywords:wholeexomesequencing?lungcancer?predisposinggenes?genemutation 一一外显子组是一个物种基因组中全部外显子区域的总和?它是基因行使其功能最直接的体现?人类外显子组序列约占人类全部基因组序列的1%?但大约包含85%的致病突变?全外显子组测序(wholeexomesequencing?WES)是一种高效的基因 组分析法?基于捕获技术的准确性和测序技术的高 通量性?将基因组中全部的外显子区域捕获富集并 进行测序?外显子组测序是一种特异性测序?单纯 针对基因组编码区域及其侧翼序列?其基本流程包 括外显子区域序列的富集二高通量测序及测序数据